相比傳統的數據儲存方式,DNA存儲具有占用空間小、獲取容易、儲存時間長3大優勢,但也面臨存儲速度慢、花費高的弊端。隨著技術的不斷成熟,它真的能成為主流嗎?
全球每年產生的數據需要4180億個1TB硬盤才能放下,若是把如此龐大的數據放到DNA上,只需 1kg DNA就夠了!DNA存儲才是未來!
幾年前,甚至研究人員也不愿使用DNA來存儲數據,因為這看起來過于科幻,并沒有任何實用價值。今天,我們可以使用正確的軟件和生物化學模塊擴展PostgreSQL,并在DNA上運行SQL。
在這個數據爆炸的時代,全球的數據不僅遠遠超過我們理解數字的能力(比如你知道Zettabytes是多少嗎?)更超過了我們的儲存能力。
一切都是數字化的,一切都越來越多地運行在基于數據訓練算法的應用程序上,而這些算法反過來會產生更多的數據來為更多的下游應用程序和算法提供信息。你懂了吧?
簡單地說,按照這種速度,很快就沒有足夠的數據存儲和計算材料可供使用。這就是為什么人們現在已經開始尋找替代的數據存儲介質的原因。使用DNA來存儲數據,這聽起來很奇怪,實際上很有意義。現在,研究人員取得了突破,使他們能夠將DNA存儲整合到流行的開源數據庫PostgreSQL中。
DNA是如何儲存數據的?
DNA的核心是數據存儲層。DNA由四種堿基組成:腺嘌呤、鳥嘌呤、胞嘧啶和胸腺嘧啶(又名AGCT)。從這四個堿基中,DNA形成由三個核苷酸組成的基團(稱為密碼子)。密碼子是給我們的細胞指示蛋白質形成的單位。
我們的信息技術基礎設施基于以位(bits)為單位的信息存儲(由兩位數字組成:0和1),而DNA信息存儲在四個潛在基本單元的串中。為了將非遺傳信息存儲在DNA中,我們必須首先將二進制數據從位轉換為DNA數據(AGCT)結構。
將Bits轉換為DNA序列
理論部分實際上相當簡單。與使用硅或磁性介質(它們的工作原理是將狀態存儲為1和0的序列)相同,存儲A、G、C和T的序列。但是,這在實踐中是如何工作的呢?如何在DNA中寫入和讀取數據呢?
這聽起來可能有點遙不可及,但分子技術的進步已經使它成為可行——盡管并不完美。
例如,微軟展示了世界上第一個自動DNA數據存儲和檢索系統。如果你想知道這個DNA來自哪里:這是合成DNA,產生它的是系統的一部分。
天然存在的DNA由兩條核苷酸鏈的雙螺旋的形式構成。相反,用于數據存儲的DNA是單鏈核苷酸序列,也稱為寡核苷酸(oligo),它通過化學過程合成,該化學過程一次構建一個核苷酸。
使用DNA在現實世界中存儲數據
倫敦帝國理工學院 SCALE實驗室負責人Heinis和Eurecom數據科學系助理教授Appuswamy 在創新數據系統研究會議上發表了題為“ OligoArchive: Using DNA in the DBMS storage hierarchy ”的研究論文。雖然他們不是第一個使用DNA存儲和檢索數據的人,但他們是第一個使用結構化數據、與現成的數據庫集成、并超越存儲、實現計算的人。
關于DNA作為數據存儲層的第一件事是,每次執行寫操作時,都必須合成寡核苷酸。這將如何在實踐中發揮作用?實驗室技術人員是否必須待命執行此操作,并“重新填充”用于化學過程的原材料?
并非如此,根據Appuswamy和Heinis的說法,這是微軟通過其自動DNA存儲和檢索系統所證明的價值所在。這表明,在沒有人參與的情況下操作這樣一個過程是可能的。就像除了維護,沒有人監督數據中心的日常運作一樣,這同樣適用于基于DNA的數據中心。
盡管如此,我們還遠沒有用合成DNA陣列取代硬盤。首先,以這種方式存儲數據的現代技術非常緩慢。剛開始,科學家們花了一周時間來存儲一兆字節的數據。
Appuswamy和Heinis一致認為,在這方面還需要更多的工作。但這超出了他們自己的研究范圍,所以只能等待生化合成過程進一步提高。
首先,他們注意到存儲速度已經越來越快,目前速度是每秒幾KB。雖然與SSD相比,這仍然非常緩慢,但這是一個很大的進步。對于Appuswamy和Heinis的研究(即歸檔存儲),這實際上是可以接受的。
數據庫引擎使用三層存儲層次結構,由具有不同價格/性能特性的設備組成。性能層存儲高性能OLTP和實時分析應用程序訪問的數據。
容量層存儲由延遲不敏感(latency-insensitive)的批分析應用程序訪問的數據。歸檔層用于存儲很少訪問的數據,例如,在安全合規性檢查或法律審計期間。今天磁帶通常用于這一層。
OligoArchive通過將基于磁帶的歸檔層替換為基于DNA的歸檔層來改變數據庫存儲層次結構。合成DNA的存儲需要額外的措施,對于普通的設備來說,基于DNA的存儲是否有效還值得懷疑。但無論如何,數據和數據庫都將進入云端,只要你的數據安全地存儲在數據中心,對終端用戶來說,這都是一個黑匣子。
在DNA上運行SQL
Appuswamy和Heinis還指出,即使它仍然很慢,DNA存儲提供了很大的并行處理潛力。因為它很豐富、價格低廉 - 或者更確切地說,我們希望它最終會是這樣。按照目前的速度,存儲一分鐘的高質量立體聲將花費100,000美元。
盡管使用合成DNA進行大規模存儲仍然過于昂貴,但Appuswamy和Heinis表示,他們預計成本會下降,這是包括存儲技術在內的每一項科技突破的典型方式。
如果合成寡核苷酸在經濟上變得可行,那么擁有大量的合成寡核苷酸將是一個合理的預期。這意味著許多DNA存儲單元可以并行運行的巨大潛力。雖然不是每種算法的每一個方面都是可并行的,但對于那些算法來說,可以實現極大的加速。這就把我們帶到了一個關鍵點。
直到今天,DNA一直被用于存儲非結構化文件,無論是文本還是視頻,或者其他什么。Appuswamy和Heinis所做的是將DNA存儲整合到關系數據庫中。他們采用了標準數據庫基準測試TPC-H中包含的數據和查詢,并在PostgreSQL實例上運行了TPC-H。不是串行訪問,而是隨意選擇數據。
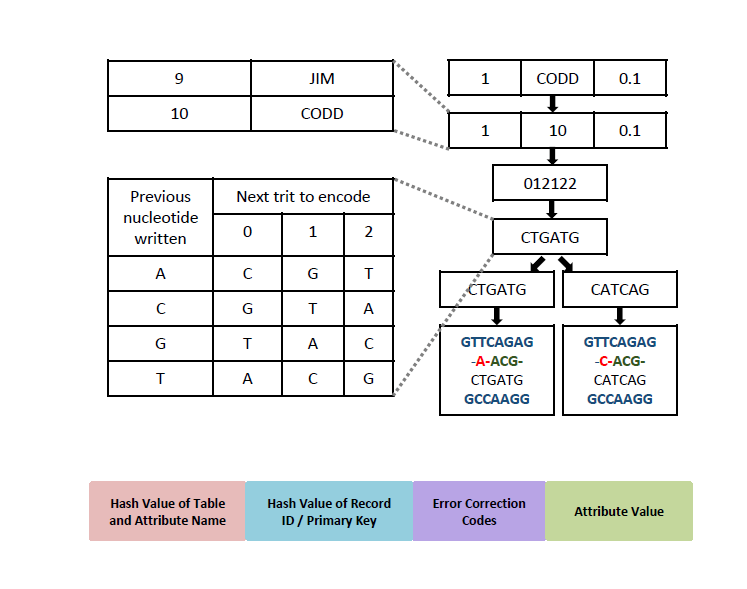
使用后端的DNA在數據庫系統中存儲結構化數據,并通過SQL查詢,這在今天已成為現實。
研究人員為PostgreSQL構建了歸檔和恢復工具(pg_oligo_dump和pg_oligo_restore),對DNA上的關系數據執行模式感知編碼和解碼,并使用這些工具將12KB TPC-H數據庫歸檔到DNA,執行in-vitro計算,以及再次恢復它。
這是巨大的。這意味著現在DNA存儲還可以支持SQL操作,來有選擇地訪問和處理部分數據。請注意,數據不會被提取到數據庫來執行操作。Appuswamy和Heinis找到了一種方法來處理寡核苷酸中的SQL連接。這超出了生化儲存的范圍——它還需要生物化學計算。
然而,要做到這一點,研究人員必須處理一系列與用于編碼和解碼DNA信息的技術缺陷有關的問題。對DNA進行操作需要專門的編碼技術,這些技術可以產生適合生化操作的寡核苷酸。讀取DNA數據目前非常容易出錯,以前的工作依賴于數據過度表示(over-representation):數據被寫在許多副本中,因此如果原始數據被破壞,備份還在。
相比之下,Appuswamy和Heinis依賴于元數據。他們利用數據庫模式感知,在編寫的塊中添加一些額外的數據位。研究表明,這可以在編碼(寫入)過程中提高密度,并有助于在解碼(讀取)過程中識別錯誤。他們注意到這比預期效果更好。
DNA是數據的未來嗎?
盡管部分技術還不成熟,但這是一個重大突破。擁有足夠的數據中心存儲空間已經成為一個game changer。但是,將像DNA這樣豐富的東西作為一種可行的存儲和計算介質的意義可能會超出我們的想象。
這可能只是朝這個方向邁出的第一步,但每一個旅程都從第一步開始,Appuswamy和Heinis并不是獨行者。
Eurecom,CNRS,ICL,UCA以及DNA合成初創企業Helixworks已獲得歐盟資助,以進一步開展DNA儲存研究。該系統將被設計成支持數據編碼的全自動循環,將其合成為DNA,并通過序列讀取數據。它將存儲各種不同的數據類型,并在存儲和精確檢索數據時實現近距離數據處理。
將數據存儲在DNA中的進一步研究將由歐盟資助
Appuswamy和Heinis提到,到目前為止,主要是其他研究人員對這個感興趣,至于說公司,微軟似乎比任何公司都表現出了更多的興趣。
掌握這項技術可能意味著擁有未來,因為這一領域的突破將產生巨大的影響。Appuswamy和Heinis指出,這表明了人們的態度:
“幾年前,人們會認為這太離譜了。今天,當我們告訴他們我們正在做什么時,他們會說——多告訴我們一些”。
-
存儲
+關注
關注
13文章
4332瀏覽量
85954 -
DNA
+關注
關注
0文章
243瀏覽量
31062
原文標題:存儲1分鐘聲音花10萬美元,DNA存儲真的有未來嗎?
文章出處:【微信號:AI_era,微信公眾號:新智元】歡迎添加關注!文章轉載請注明出處。
發布評論請先 登錄
相關推薦




 使用DNA在現實世界中存儲數據
使用DNA在現實世界中存儲數據










評論