NVIDIA 數據科學家本周在享有盛譽的MICCAI 2021醫學成像會議上,在腦腫瘤分割挑戰驗證階段占據了前 10 名的三位。
現在已經進入第十個年頭, BraTS 挑戰項目要求申請者提交最先進的人工智能模型,用于在多參數磁共振成像( mpMRI )研究中分割異質性腦膠質母細胞瘤亞區域,這是一項極具挑戰性的任務。
參與者還可以關注分類方法的第二項任務,以預測 MGMT 啟動子甲基化狀態。
2000 多個 AI 模型被提交給了挑戰,該挑戰由醫學圖像計算和計算機輔助干預學會、北美放射學會和美國神經放射學會聯合組織。
NVIDIA 開發者在挑戰驗證階段排名第一、第二和第七,每個人都創建了不同類型的用于腫瘤分割的 AI 模型方法,包括優化的 U-Net 模型、具有自動超參數優化的 SegResNet 模型和基于 transformer 的計算機視覺方法的 Swin-UNETR 模型。
NVIDIA 獲獎者都使用了開源 PyTorch 框架MONAI(人工智能醫療開放網絡),這是一個由學術界和行業領袖構建的免費、社區支持的計劃,旨在將醫療成像深度學習的最佳實踐標準化。
用于腦腫瘤分割的優化 U-Net –排名# 1
該優化 DU 網絡模型是一種編碼器 – 解碼器類型的卷積網絡體系結構,用于快速、精確的圖像分割,它首先處于 BraTS 驗證階段。它的標準化統計排名得分為 0 。 267 。
設計優化 U 型網絡的起點是 BraTS 2020 獲獎解決方案:神經網絡在腦腫瘤分割中的應用。該團隊的目標是優化 U-Net 體系結構以及培訓計劃。為了找到最佳的模型結構,數據科學家進行了廣泛的燒蝕研究,發現香草 U-Net 和深度監督產生了最好的結果。
進一步優化 U-Net 模型,在輸入端添加額外通道,對前景體素進行一次熱編碼,將編碼器深度和卷積通道數量增加一個級別。通過在較低的解碼器級別上添加兩個額外的輸出頭,可以更好地實現梯度流和更準確的預測,從而在深度監督下更好更快地訓練模型。MONAI用于數據預處理,以清除數據并去除數據中的噪聲,以及推斷模型。 NVIDIA 數據加載庫( DALI )使用并支持數據擴充,這是一種用于人為擴大數據集大小的技術,它通過將數據擴充卸載到 GPU 來解決 CPU 瓶頸問題。
帶有最新 PyTorch、cuDNN 和 CUDA 版本的 NVIDIA PyTorch 容器用于優化 U-Net 模型以進行快速訓練。自動混合精度 (AMP) 用于將 AI 模型的內存占用減少 2 倍并加快訓練速度。訓練在 8 個 NVIDIA A100 GPU 上完成 1000 個 epoch,與 V100 GPU 相比,速度提高了 2 倍。幾乎 100% 的 GPU 被使用,展示了網絡優化以高效使用 GPU。此 3D U-Net 模型可用于任何 3D 模式,例如 MRI 和 CT。了解更多 GPU 高效的 nnU-Net 實現。

圖 1 。挑戰驗證數據集上的預測。在第一行, T2 模態是可視化的。第二行顯示以下顏色的模型預測:紫色 – 背景、藍色 -NCR 、綠松石色 -ED 、黃色 -ET 。
SegResNet : 3D 大腦磁共振成像語義分割中的冗余減少–排名# 2
該方法在 BRAT 挑戰中排名第二,將在 MONAI 中提供,基于 MONAI 組件,旨在展示其應用的實用性和靈活性。主要模型是 MONAI 中的 SegResNet 架構,這是一種基于標準編碼器 – 解碼器的卷積神經網絡( CNN ),類似于 U-Net 。該方法是 MONAI 自動化( AutoML )計劃的一部分,使用超參數優化和調優自動選擇超參數。
該方法在驗證階段的排行榜( team NVAUTO )中取得了最佳性能,并在基于個案排名和擾動分析的綜合排名中取得了 0 。 272 的排名。組織者表示,該排名與第一排名的解決方案在統計上沒有顯著差異,兩種方法被認為在統計上相似。
這項工作增加了兩項新貢獻,以進一步提高性能。首先,對訓練過程進行修改,以在學習的特征表示上強制執行某些屬性。通過借鑒自監督文獻中的思想,將特征維度正則化,使不同解剖區域之間的冗余度最小。同時,相同解剖結構的區域被鼓勵相似。這允許更好的網絡行為和泛化。其次,采用自適應融合技術自適應地選擇模型子集進行融合。這有助于避免某些模型預測中的潛在異常值,并進一步提高最終的集合性能。
該方法在 MONAI 中實現,以 PyTorch 為基礎,并在四個 NVIDIA V100 GPU 上使用骰子損失函數進行了為期 16 小時的 300 個歷元的訓練。該團隊使用 NVIDIA 提供的 PyTorch 容器和 AMP 在超參數優化過程中實現快速訓練。該方法通過 5 倍交叉驗證進行訓練,并從每個折疊中保留表現最佳的檢查點。總的來說,保存了 25 個模型檢查點,但使用自適應加密,只有一半用于最終預測。由于該方法是完全基于 CNN 的,推理時間很快,并且可以在整個輸入圖像上一步完成,無需任何滑動窗口。單模型推理只需不到一秒鐘的時間,這使得高通量和幾乎實時的結果成為可能,這在臨床環境中非常重要。
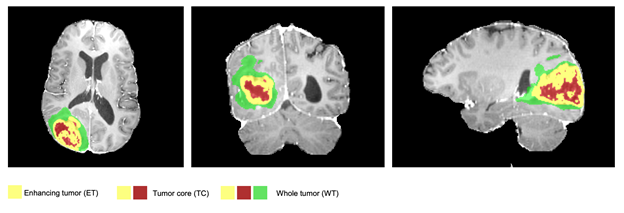
圖 2 。一個典型的分割示例,預測的標簽覆蓋在 MRI 軸向、矢狀面和冠狀面切片上。整個腫瘤( WT )類別包括所有
可見標簽(綠色、黃色和紅色標簽的結合),腫瘤核心( TC )類別為
紅色和黃色的結合,增強腫瘤核心( ET )級別顯示為黃色
(過度活躍的腫瘤部分)。
Swin UNETR :用于腦腫瘤三維語義分割的移位窗口 transformers –排名第 7
在 BraTS 挑戰賽中排名第七的 Swin UNETR 是一個基于 transformer 的模型,而不是 CNN 模型。在 MONAI 中實現,它在整個腫瘤、腫瘤核心和增強腫瘤分割類中的平均 Dice 得分為 92 。 94% , Ha USD orff 距離為 1 。 7 。
transformers 是一類新的基于深度學習的模型,用于序列到序列的預測任務。在其原始公式中,它們由編碼器和解碼器組成。編碼器由多個層次組成,多個層次的感知器跟隨多個層次的自我注意。每個模塊的輸入通過剩余塊添加到輸出,并進行歸一化。自我注意層學習從隱藏層計算的值的加權和,可以突出給定輸入序列的重要特征。雖然它們最初被提議用于自然語言處理中的機器翻譯任務,但后來它們已成功應用于計算機視覺和蛋白質藥物生成等其他領域,并在各種基準測試中取得了最先進的性能。在計算機視覺中, transformer s 在各種基準方面取得了新的最先進的表現。 Swin UNETR 是一種利用 GPU 體系結構和性能不斷進步來構建模型的新方法。
Swin transformers 是分層 transformer s ,其表示是使用移位窗口( Swin )計算的。這些 transformer 非常適合計算機視覺任務,如目標檢測、圖像分類、語義分割等。 Swin transformers 可以更有效地模擬兩個域之間的差異,例如對象比例的變化和圖像中像素的高分辨率,并且可以作為通用的視覺管道。該 NVIDIA SWN UNETR 模型利用 SWN transformer 編碼器直接利用輸入數據的 3D 補丁,而不依賴 CNN 進行特征提取。這使得 Swin UNETR 能夠訪問輸入數據中的上下文多模態信息,并將其作為標記化嵌入 transformer 編碼器進行有效處理。然后,基于 transformer 的 Swin UNETR 編碼器通過跳過連接連接到 U 形架構中的 CNN 解碼器,以進行最終分段預測。
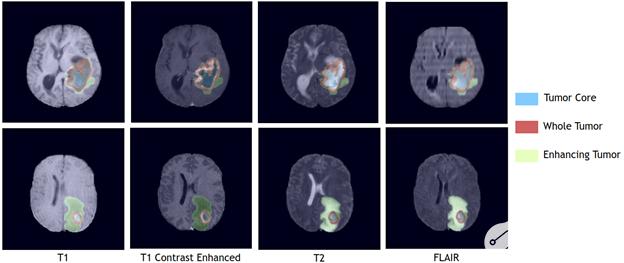
圖 3 。使用 BraTS2021 驗證數據集,覆蓋在 4 種單獨成像模式上的 TC 、 WT 和 ET 區域的 Swin UNETR 分割輸出。
NVIDIA 的 SWI UNETR 模型在 NVIDIA DGX-1 團簇上使用八個 GPU 進行訓練,初始學習率為 0 。 0008 ,并使用 AdamW 優化算法。使用 128 * 128 * 128 輸入數據的隨機面片以及隨機軸鏡像翻轉和強度偏移的數據增強策略。 每輪培訓需要 24 小時才能完成。與常用的基于 CNN 的分割模型相比, Swin UNETR 在失敗次數方面更有效,在可訓練參數數量方面具有中等的模型復雜度。它可以被有效地訓練和用于推理。對于模型優化,使用一個通用的軟骰子損失函數學習分割不同的腦腫瘤區域,每個類有一個單獨的輸出通道。在整個 BRATS21 訓練集上使用 5 倍交叉驗證方案對模型進行訓練,其中通過平均 2 個不同 5 倍交叉驗證的 10 個模型的輸出來計算最終分割輸出。
從 MONAI 開始。
在這里查找 BRAT 參與者的完整排行榜。

關于作者
Vanessa Braunstein 在 NVIDIA 的醫療團隊從事產品營銷工作。此前,她在基因組學、醫學成像、制藥、化學和診斷公司從事產品開發和營銷。她學習分子和細胞生物學、公共衛生和商業。
審核編輯:郭婷
-
NVIDIA
+關注
關注
14文章
5240瀏覽量
105768 -
gpu
+關注
關注
28文章
4910瀏覽量
130652
發布評論請先 登錄
NVIDIA在多模態生成式AI領域的突破性進展
深蘭科技醫療大模型榮獲MedBench評測第一
東軟醫療大模型覆蓋眾多應用場景
AI在醫療健康和生命科學中的發展現狀
英偉達GTC2025亮點:NVIDIA與GE醫療合作 引入物理AI推進自主診斷成像開發
NVIDIA大語言模型在推薦系統中的應用實踐



















評論