使用 NVIDIA Clara 構建的醫療成像 AI 模型現在可以在云計算的 MD.ai 上本地運行,從而使用現代 web 瀏覽器實現協作模型驗證和快速注釋項目。這些 NVIDIA Clara 模型可免費用于任何 MD.ai 合作研究項目,如器官或腫瘤分割。
人工智能解決方案已被證明有助于簡化放射學和企業成像工作流程。然而,創建、共享、測試和縮放計算機視覺模型的過程并不像所有模式、條件和結果那樣簡化。需要幾個關鍵組件來創建穩健的模型,并支持最多樣化的采集設備和患者群體。這些關鍵組件可以包括為未注成像研究創建基本事實的能力,以及在全球范圍內合作評估模型與驗證數據的使用的能力。
MD.ai 的實時協作標注平臺和英偉達 Clara 深度學習培訓框架有助于創建更健壯的模型構建和協作。
在這篇文章中,我們將介紹 Clara Train MMAR 的基礎知識,以及準備使用 MD.ai 所需的步驟。只需幾個步驟,您就可以在 MD.ai 上部署這些預訓練模型中的任何一個,以實現無縫的基于 web 的評估和協作。在 MD.ai 上部署之后,這些模型可以用于任何現有或新的 MD.ai 項目。
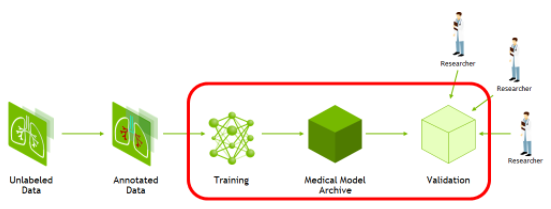
圖 1 。培訓和驗證 AI 模型所需的工作流程 。
NVIDIA Clara Train
Clara 訓練框架是一個基于 Python 的應用程序包 NVIDIA Clara Train SDK. 該框架旨在基于 NVIDIA 研究人員內部構建的優化、即用、預訓練的醫學成像模型,在醫學成像領域快速實施深度學習解決方案。
Clara 培訓框架使用模型的標準結構 醫學模型檔案 ( MMAR ),其中包含預培訓模型以及定義用于培訓、微調、驗證和推理的端到端開發工作流的腳本。
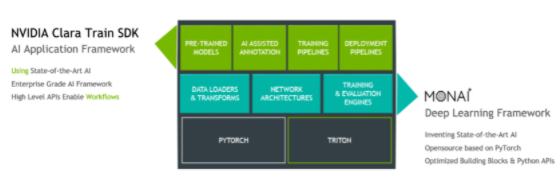
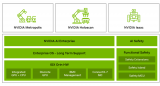
圖 2.Clara Train SDK 和 MONAI 深度學習框架高級架構。
Clara Train v4.0 + SDK 使用基于組件的體系結構,該體系結構構建于基于 PyTorch 的開源框架 MONAI (人工智能醫療開放網絡) 。 MONAI 提供了醫療成像領域優化的基礎功能,可用于在本機 PyTorch 范式中構建培訓工作流。 Clara Train SDK 使用這些基礎組件(如優化的數據加載程序、轉換、丟失函數、優化器和度量)來實現打包為 MMAR 的端到端培訓工作流。
MD.ai
MD.ai 提供了一個基于 web 的云本地注釋平臺,支持臨床醫生和研究人員團隊之間通過共享工作空間進行實時協作。您還可以加載多個深度學習模型以進行實時評估。
該平臺為數據集構建和 AI 項目創建提供了一個簡單無縫的界面。它為用戶提供了一套廣泛的工具,用于注釋數據和構建機器學習算法,以加速人工智能在醫學中的應用,特別關注醫學成像。
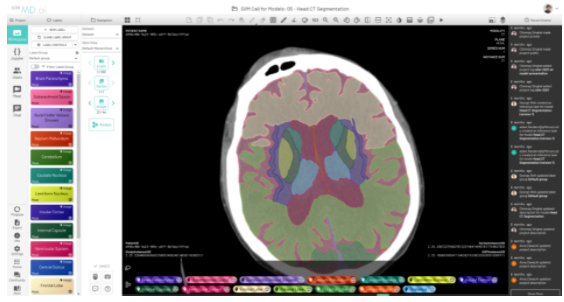
圖 3 。顯示大腦分割的 MD.ai 用戶界面 。
將此功能與在 MD.ai 平臺上快速部署 Clara 訓練模型 MMAR 的能力相結合,可以為您提供一個端到端的工作流,該工作流涵蓋快速模型開發、模型訓練、微調、推理以及快速評估和可視化。這種端到端的能力簡化了模型從研發到生產的過程。
解決方案概述
Clara Train 的起點是NGC Clara Train 采集。 在這里,您可以找到 Clara Train SDK 容器、一組免費提供的、經過預訓練的模型,以及一組 Jupyter 筆記本電腦,其中介紹了 SDK 的主要概念。所有 Clara Train 模型共享前面提到的 MMAR 格式。
Clara Train MMAR 定義了一個標準結構,用于存儲定義模型開發工作流所需的文件,以及執行模型進行驗證和推斷時生成的文件。該結構定義如下:
ROOT config config_train.json config_finetune.json config_inference.json config_validation.json config_validation_ckpt.json environment.json commands set_env.sh train.sh train with single GPU train_multi_gpu.sh train with 2 GPUs finetune.sh transfer learning with CKPT infer.sh inference with TS model validate.sh validate with TS model validate_ckpt.sh validate with CKPT validate_multi_gpu.sh validate with TS model on 2 GPUs validate_multi_gpu_ckpt.sh validate with CKPT on 2 GPUs export.sh export CKPT to TS model resources log.config ... docs license.txt Readme.md ... models model.pt model.ts final_model.pt eval all evaluation outputs: segmentation / classification results metrics reports, etc.
所有提供用于 Clara Train 的預訓練模型,以及使用 Clara Train 框架開發的自定義模型都使用此結構。為了準備與 MD.ai 一起使用的 MMAR ,我們假設一個預先訓練的模型,并將重點放在部署的幾個關鍵組件上。
第一個組件是 environment.json 文件,它定義了模型的公共參數,包括數據集路徑和模型檢查點。例如, Clara 序列分段任務中的 environment.json 文件定義了以下參數:
{ "DATA_ROOT": "/workspace/data/Task09_Spleen_nii", "DATASET_JSON": "/workspace/data/Task09_Spleen_nii/dataset_0.json", "PROCESSING_TASK": "segmentation", "MMAR_EVAL_OUTPUT_PATH": "eval", "MMAR_CKPT_DIR": "models", "MMAR_CKPT": "models/model.pt" "MMAR_TORCHSCRIPT": "models/model.ts"
}
準備與 MD.ai 集成的模型時,請確保 MMAR 的models/目錄中包含經過培訓的MMAR_CKPT和MMAR_TORCHSCRIPT。它們分別通過執行捆綁的train.sh和export.sh生成。
-
train.sh腳本執行模型訓練,這需要輸入數據集使用DATA_ROOT和DATASET_JSON,并生成 MMAR _ CKPT 。 -
infer.sh腳本將此檢查點序列化到用于推斷的MMAR_TORCHSCRIPT中。
對于預訓練模型,提供了檢查點和 TorchScript ,您可以關注推理管道。使用 MMAR 的infer.sh腳本執行推斷:
1 #!/usr/bin/env bash 2 my_dir="$(dirname "$0")" 3 . $my_dir/set_env.sh 4 echo "MMAR_ROOT set to $MMAR_ROOT" 5 6 CONFIG_FILE=config/config_validation.json 7 ENVIRONMENT_FILE=config/environment.json 8 python3 -u -m medl.apps.evaluate \ 9 -m $MMAR_ROOT \ 10 -c $CONFIG_FILE \ 11 -e $ENVIRONMENT_FILE \ 12 --set \ 13 DATASET_JSON=$MMAR_ROOT/config/dataset_0.json \ 14 output_infer_result=true \ 15 do_validation=false
此腳本對environment.json中定義的完整數據集的config_validation.json中定義的驗證子集進行推斷。如果參考測試數據隨 MMAR 一起提供,則必須定義該數據的路徑。集成 MMAR 時, MD.ai 直接處理數據集,這些值作為集成的一部分被覆蓋。
要在 MD.AI 上部署您自己的預訓練 AI 模型進行推理,您必須已經有一個現有項目或在平臺上創建一個新項目。項目還必須包含用于測試模型的數據集。有關更多信息,請參閱 立項 。
接下來,要部署 AI 模型,必須將推理代碼轉換為與平臺兼容的特定格式。以下文件是成功部署的最低要求:
config.yaml
mdai_deploy.py
requirements.txt
model-weights
對于 NVIDIA Clara 型號,我們已為您進一步簡化了此功能,無需從頭開始編寫這些文件。我們為 NGC 目錄支持的每個不同類別的深度學習模型提供了框架代碼:分類、分段等。您可以下載特定于模型的框架代碼,進行一些調整,這些調整將在本文后面介紹,然后將模型上傳到 MD.ai 上進行推斷。
推理步驟
準備好 MMAR 后,下面介紹如何直接使用它在 MD.ai 上運行模型。這篇文章將引導您了解一個已經部署在平臺上的示例細分模型 在 MD.ai 上運行分段模型的框架代碼 ,它實際上是 NVIDIA 的 CT spleen segmentation 模型的代碼。
現在,要使用相同的 MMAR 格式部署 肝臟和腫瘤分割 模型,請執行以下步驟:
- 下載分割模型的框架代碼。
- 從 NGC 目錄下載用于肝臟和腫瘤分割模型的 MMAR。
-
在下載的框架代碼中,用下載的 MMAR 文件夾替換
/workspace/clara_pt_spleen_ct_segmentation_1文件夾。 -
在
/workspace/config_mdai.json文件中,進行以下更改:
1 {
2 “type” : “segmentation”,
3 “root_folder”: “clara_pt_spleen_ct_segmentation_1”,
4 “out_classes” : 2,
5 “data_list_key”: “test”
6 }
- root_folder– 將此鍵值替換為下載的 MMAR 文件夾的名稱,例如肝臟和腫瘤示例的 clara _ pt _ liver _和_ tumor _ ct _ segmentation _ 1 。
- out_classes– 將此值替換為模型的輸出類數,如本例中的 3 (背景: 0 、肝臟: 1 和腫瘤: 2 )。
- data_list_key– 替換為 MMAR 的 config / config _ interference.json 文件的 data _ list _ key 屬性中提到的密鑰名稱,例如 testing 。
-
在
/mdai文件夾中,進行以下更改:
-
在
config.yaml文件,中,將clara_version鍵更改為您的模型使用的適當版本(例如, 3.1.01 或 4.0 )。
1 base_image: nvidia 2 clara_version: 4.0 3 device_type: gpu
- 在[EZX29 ],中添加了任何額外的依賴項,比英偉達 Clara 基本圖像和文件中已經存在的那些依賴性要多。
這將為您的模型在 MD.ai 上的部署做好準備。脾臟和肝臟腫瘤分割模型都已部署在 MD.ai 平臺上,并且是 可供評估 。
分類模型也可以執行類似的步驟,盡管我們正在努力進一步簡化這種集成。有關將胸部 X 光分為 15 種異常的示例 NVIDIA 模型的骨架代碼,請參見 GitHub 上的 示例:用于胸部 x 光疾病分類的 NVIDIA MMAR 。該型號也是 部署在公共站點上 。
當代碼準備就緒時,必須將其包裝在 zip 文件中,以便將其上載到 MD.ai 上進行推斷。
您的模型現在可以在 MD.ai 中對您選擇的任何數據集進行嘗試了!
最好的是,所有模型只需在MD.ai平臺上部署一次。一旦NVIDIA Clara 模型部署到MD.ai上,它就可以通過使用模型克隆功能在任何MD.ai項目中使用。下面是一個例子,通過復制[EZX37 ]值,將英偉達肝分割模型克隆為一個新的MD AI項目:
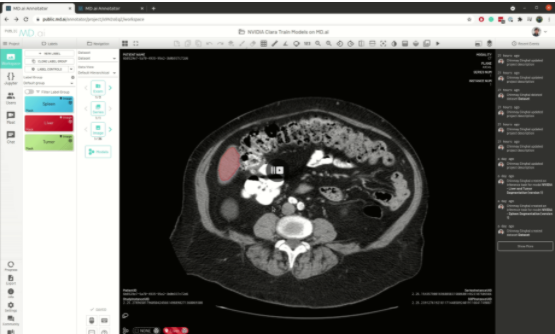 圖 5 。基于 MD.ai 的模型克隆工作流.
圖 5 。基于 MD.ai 的模型克隆工作流.未來特征
我們正致力于簡化集成,以盡量減少部署 MMAR 所需的步驟,并計劃消除所有代碼修改,以便只需單擊一個按鈕即可輕松部署。
MD.ai 計劃預先部署 NGC 上可用的所有模型,以便您可以通過從我們的公共項目克隆直接使用它們,從而避免您自己部署 MMAR 的過程。我們還將創建 NVIDIA Clara 入門包,這樣您就可以輕松地開始使用預裝到項目中的選定型號。
另一個重要的計劃是增加對在 MD.AI 上訓練 AI 模型的支持。當我們有了這個平臺,您可以有效地使用英偉達 AI 輔助注釋產品在平臺上幫助用戶注釋得更快更容易,而不是從頭開始。
總結
在本文中,我們重點介紹了每個平臺的關鍵組件以及在 MD.ai 上快速部署使用 NVIDIA Clara 構建的醫療成像模型所需的步驟。
關于作者
Chinmay Singhal 是 MD.ai 的機器學習工程師。他的工作圍繞 MD.ai 機器學習模型部署平臺的設計和開發展開。他在紐約大學獲得了數據科學碩士學位,專注于深度學習。
Leon Chen 是 MD.ai 的聯合創始人兼首席執行官,負責構建醫療 ai 的未來 IDE 。他是哈佛大學培訓的內科醫生、 Kaggle 碩士、 Keras.js 等流行開源軟件的創造者和電子音樂家。
Kris Kersten 是 NVIDIA 的解決方案架構師,專注于 AI ,致力于擴展 ML 和 DL 解決方案,以解決當今醫療領域最緊迫的問題。在加入 NVIDIA 之前, Kris 曾在 Cray 超級計算機公司工作,研究從低級緩存基準測試到大規模并行模擬的硬件和軟件性能特征。
審核編輯:郭婷
-
NVIDIA
+關注
關注
14文章
4978瀏覽量
102993 -
云計算
+關注
關注
39文章
7774瀏覽量
137355 -
AI
+關注
關注
87文章
30734瀏覽量
268895
發布評論請先 登錄
相關推薦
NVIDIA推出全新生成式AI模型Fugatto
使用NVIDIA AI平臺確保醫療數據安全
NVIDIA AI助力實現更好的癌癥檢測
NVIDIA RTX AI套件簡化AI驅動的應用開發
NVIDIA AI Foundry 為全球企業打造自定義 Llama 3.1 生成式 AI 模型

揭秘NVIDIA AI Workbench 如何助力應用開發
NVIDIA 通過 Holoscan 為 NVIDIA IGX 提供企業軟件支持,實現邊緣實時醫療、工業和科學 AI 應用

STM CUBE AI錯誤導入onnx模型報錯的原因?
NVIDIA的專用AI平臺如何推動下一代醫療健康行業的發展
NVIDIA在加速識因智能AI大模型落地應用方面的重要作用介紹
使用cube-AI分析模型時報錯的原因有哪些?
NanoEdge AI的技術原理、應用場景及優勢






 NVIDIA Clara醫療成像AI模型在MD.ai項目中的應用
NVIDIA Clara醫療成像AI模型在MD.ai項目中的應用










評論