本文介紹了一種從稀疏標注的體積圖像中學習的體積分割網絡。我們概述了該方法的兩個有吸引力的用例:(1)在半自動設置中,用戶注釋要分割的體積中的一些切片。網絡從這些稀疏注釋中學習并提供密集的3D分割。(2)在全自動設置中,我們假設存在一個有代表性的、稀疏注釋的訓練集。在這個數據集上訓練,網絡密集分割新的體積圖像。所提出的網絡擴展了Ronneberger等人先前的u-net架構。通過將所有2D操作替換為對應的3D操作。該實現在訓練期間執行動態彈性變形以實現有效的數據增強。它是從頭開始端到端訓練的,即不需要預先訓練的網絡。我們在復雜、高度可變的3D結構(非洲爪蟾腎臟)上測試了所提出方法的性能,并在兩個用例中都取得了良好的效果。
「關鍵詞:」卷積神經網絡,3D,生物醫學體積圖像分割,非洲爪蟾腎臟,半自動,全自動,稀疏標注
1 Introduction
體積數據在生物醫學數據分析中非常豐富。使用分段標簽對此類數據進行注釋會造成困難,因為計算機屏幕上只能顯示2D切片。因此,以逐片的方式注釋大量數據是非常繁瑣的。這也是低效的,因為相鄰切片顯示幾乎相同的信息。特別是對于需要大量注釋數據的基于學習的方法,3D 體積的完整注釋不是創建能夠很好地泛化的大型和豐富的訓練數據集的有效方法。
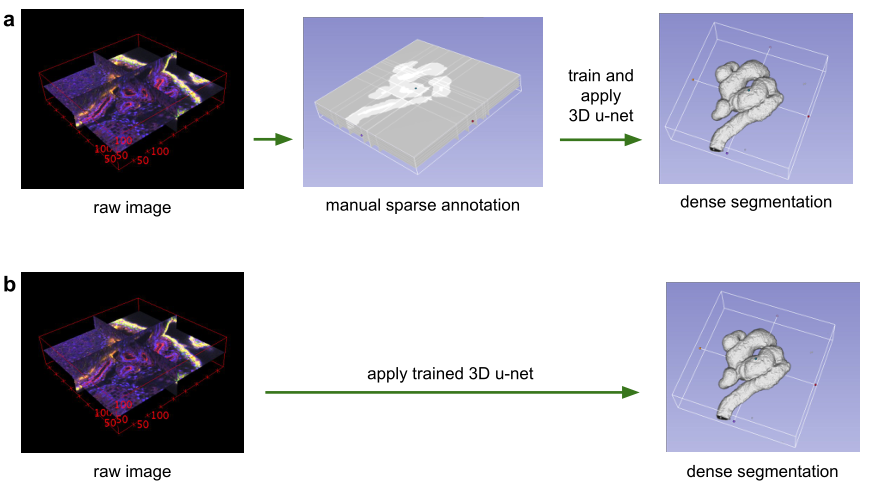
圖 1:使用3D u-net進行體積分割的應用場景。(a)半自動分割:用戶注釋要分割的每個體積的一些切片。網絡預測密集分割。(b)全自動分割:網絡使用來自代表性訓練集的帶注釋切片進行訓練,并且可以在非注釋卷上運行。
?
在本文中,我們提出了一個深度網絡,它可以學習生成密集的體積分割,但只需要一些帶注釋的2D切片進行訓練。該網絡可以以兩種不同的方式使用,如圖1所示:第一個應用案例僅針對密集化稀疏注釋的數據集;第二個從多個稀疏注釋的數據集中學習以推廣到新數據。這兩個案例都具有高度相關性。
該網絡基于之前的u-net架構,由一個用于分析整個圖像的收縮編碼器部分和一個用于產生全分辨率分割的連續擴展解碼器部分組成 [11]。雖然u-net是一個完全2D的架構,但本文提出的網絡將3D卷作為輸入,并使用相應的3D操作對其進行處理,特別是3D convolutions,3D max pooling, 和3D up-convolutional層。此外,我們避免了網絡架構[13]中的瓶頸,并使用批量歸一化[4]來加快收斂速度。
在許多生物醫學應用中,只需要很少的圖像來訓練一個泛化能力相當好的網絡。這是因為每個圖像已經包含具有相應變化的重復結構。在體積圖像中,這種效果更加明顯,因此我們可以只在兩個體積圖像上訓練一個網絡,以便推廣到第三個。加權損失函數和特殊的數據增強使我們能夠僅使用少量手動注釋切片來訓練網絡,即來自稀疏注釋的訓練數據。
我們展示了所提出的方法在困難的爪蟾腎共焦顯微數據集上的成功應用。在其發展過程中,非洲爪蟾腎臟形成了一個復雜的結構[7],這限制了預定義參數模型的適用性。首先,我們提供定性結果來證明少數帶注釋切片的稠密化質量。這些結果得到定量評估的支持。我們還提供了實驗,顯示了帶注釋的切片數量對我們網絡性能的影響。基于Caffe[5]的網絡實現作為開源提供。
1.1 Related Work
具有挑戰性的生物醫學2D圖像可以通過CNN以接近人類表現的精度進行分割[11,12,3]。由于這一成功,已經進行了幾次嘗試將3D CNNs應用于生物醫學體積數據。Milletari等人[9]提出了一種結合Hough投票法的CNN用于3D分割。然而,他們的方法不是端到端的,只適用于緊湊的團狀結構。Kleesiek等人[6]的方法是用于3D分割的少數端到端3D CNN方法之一。然而,它們的網絡并不深,在第一次卷積后只有一個最大池;因此,它不能在多個尺度上分析結構。我們的工作基于2D u-net [11],該網絡在2015年贏得了多項國際分割和跟蹤比賽。u-net的體系結構和數據擴充允許僅從幾個帶注釋的樣本中學習具有非常好的泛化性能的模型。它利用了這樣一個事實,即適當應用的剛性變換和輕微的彈性變形仍然會產生生物上似是而非的圖像。上行卷積架構,如用于語義分割的全卷積網絡[8]和u-net,仍然沒有廣泛傳播,我們只知道一種將這種架構推廣到3D的嘗試[14]。在Tran等人的這項工作中,該架構被應用于視頻,并且完整的注釋可用于訓練。本文的重點是,由于其無縫拼接策略,它可以在稀疏標注的卷上從頭開始訓練,并且可以在任意大的卷上工作。
2 Network Architecture
圖2說明了網絡架構。像標準的u-net一樣,它有一個分析和綜合路徑,每個路徑有四個解析步驟。在分析路徑中,每一層包含兩個3 × 3 × 3卷積,每個卷積后跟一個整流線性單元(ReLu),然后是一個2 × 2 × 2最大池化,每個維度的步長為2。在合成路徑中,每一層都包括一個2 × 2 × 2的上卷積,每個維度上的步長為2,然后是兩個3 × 3 × 3的卷積,每個卷積之后是一個ReLu。分析路徑中相同分辨率層的快捷連接為合成路徑提供了基本的高分辨率特征。在最后一層中,1×1×1卷積將輸出通道的數量減少到標簽的數量,在本例中為3個。該架構共有19069955個參數。正如[13]中所建議的,我們通過在最大池化之前將通道數量翻倍來避免瓶頸。我們在合成路徑中也采用這種方案。
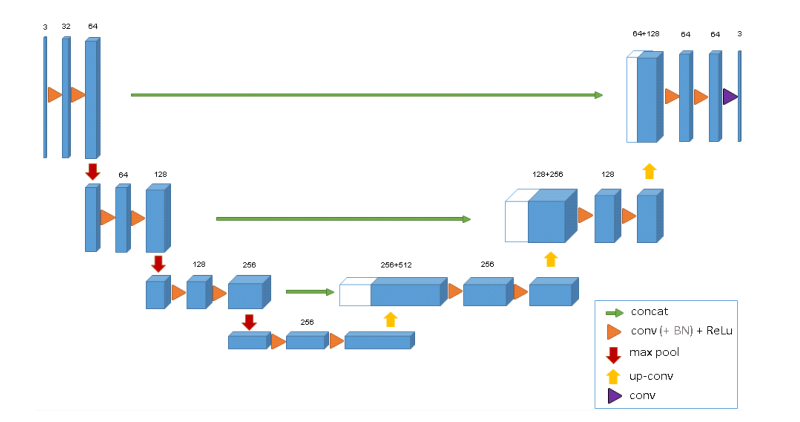
圖 2: 3D u-net架構。藍框代表要素地圖。通道的數量在每個特征圖上標出。
?
網絡的輸入是具有3個通道的圖像的132 × 132 × 116體素塊。我們在最后一層的輸出分別是x、y和z方向的44×44×28個體素。在體素尺寸為1.76×1.76×2.04的情況下,對于預測分割中的每個體素,近似感受野變為155×155×180。因此,每個輸出體素都可以訪問足夠的上下文來有效地學習。 我們還在每次ReLU之前引入了批處理歸一化(“BN”)。在[4]中,每批在訓練期間用其平均值和標準偏差進行歸一化,并使用這些值更新全局統計數據。接下來是一個層,用于顯式學習比例和偏差。在測試時,通過這些計算出的全局統計數據和學習到的標度和偏差來進行標準化。然而,我們有一個批量和幾個樣品。在這樣的應用程序中,在測試時使用當前的統計數據效果最好。 該架構的重要部分是加權的softmax損失函數,它允許我們對稀疏注釋進行訓練。將未標記像素的權重設置為零使得可以僅從標記的像素中學習,從而推廣到整個體積。
3 Implementation Details
3.1 Data
我們有三個處于Nieuwkoop-Faber階段36-37的爪蟾腎胚胎樣本[10]。其中一個如圖1所示(左)。使用配備有Plan-Apochromat 40x/1.3油浸物鏡的Zeiss LSM 510 DUO倒置共焦顯微鏡,在具有3個通道的四個瓷磚中記錄了3D數據,體素尺寸為0.88×0.88×1.02。第一個通道顯示番茄凝集素與488nm激發波長的熒光素偶聯。第二個通道顯示在405nm激發下DAPI染色的細胞核。第三個通道顯示Beta-Catenin使用標記有Cy3的二抗在564nm激發下標記細胞膜。我們使用Slicer3D在每個體積中手動注釋一些正交的xy、xz和yz切片[2]。根據良好的數據表示選擇注釋位置,即在所有3個維度上盡可能均勻地采樣注釋切片。不同的結構被賦予標簽0:“小管內”;1:“小管”;2:“背景”,3:“未標注”。未標記切片中的所有體素也獲得標記3(“未標記”)。我們對原始分辨率的下采樣版本進行了所有的實驗,在每個維度上的因子為2。因此,對于我們的樣本1、2和3,實驗中使用的數據大小在x × y × z維度上分別為248 × 244 × 64、245 × 244 × 56和246 × 244 × 59。對于樣本1、2和3,正交(yz、xz、xy)切片中人工注釋的切片數量分別為(7、5、21)、(6、7、12)和(4、5、10)。
3.2 Training
除了旋轉、縮放和灰度值增強之外,我們還在數據和ground truth標簽上應用了平滑的密集變形場。為此,我們在每個方向上間隔32個體素的網格中從標準偏差為4的正態分布中采樣隨機向量,然后應用B-spline插值。使用帶有加權交叉熵損失的softmax比較網絡輸出和ground truth標簽,我們減少常見背景的權重并增加內小管的權重,以達到小管和背景體素對損失的平衡影響。標簽為3(“未標記”)的體素對損失計算沒有貢獻,即權重0。我們使用Caffe [5]框架的隨機梯度下降求解器進行網絡訓練。為了能夠訓練大型3D網絡,我們使用了內存高效的卷積層實現。數據增強是即時完成的,這會產生與訓練迭代一樣多的不同圖像。我們在NVIDIA TitanX GPU上運行了70000次訓練迭代,大約耗時3天。
4 Conclusion
我們引入了一種端到端的學習方法,可以半自動和全自動地從稀疏注釋中分割出3D體積。它為非洲爪蟾腎臟的高度可變結構提供了準確的分割。我們在半自動設置的3折交叉驗證實驗中實現了0.863的平均IoU。在全自動設置中,我們展示了3D架構相對于等效2D實現的性能增益。該網絡是從頭開始訓練的,并且沒有針對此應用進行任何優化。我們預計它將適用于許多其他生物醫學體積分割任務。它的實現是作為開源提供的。
-
3D
+關注
關注
9文章
2875瀏覽量
107488 -
2D
+關注
關注
0文章
64瀏覽量
15198 -
圖像分割
+關注
關注
4文章
182瀏覽量
17995
原文標題:3D U-Net:從稀疏注釋中學習密集的體積分割
文章出處:【微信號:3D視覺工坊,微信公眾號:3D視覺工坊】歡迎添加關注!文章轉載請注明出處。
發布評論請先 登錄
相關推薦
針對顯示屏的2D/3D觸摸與手勢開發工具包DV102014
如何同時獲取2d圖像序列和相應的3d點云?
2D到3D視頻自動轉換系統




 3D架構相對于等效2D實現的性能增益
3D架構相對于等效2D實現的性能增益












評論