Google今天推出了一個(gè)名叫DeepVariant的開源工具,用深度神經(jīng)網(wǎng)絡(luò)來從DNA測序數(shù)據(jù)中快速精確識別堿基變異位點(diǎn)。
學(xué)科研究的革命性進(jìn)展,特別是基因?qū)W上,需要依賴于新技術(shù)的出現(xiàn)。比如桑格發(fā)明了測序法之后,才實(shí)現(xiàn)了人類基因組的測序。
再比如DNA(微陣列)芯片技術(shù)的誕生,使得大規(guī)模的基因測序成為可能。這些技術(shù)讓我們能夠獲得大量遺傳信息,可以更廣泛地應(yīng)用于健康、農(nóng)業(yè)和生態(tài)上。
基因測序領(lǐng)域里,最革命性的技術(shù)當(dāng)屬2000年初首次商用的高通量測序(縮寫為HTS)了。HTS可以大規(guī)模、低成本、快速地獲得任何生物的基因序列。
不過,HTS有個(gè)致命的問題在于,測序出來的結(jié)果不是完整的,而是碎片化的片段信息。
比如測的是人的基因序列的話(也就是說,信息量級為23對染色體上的30億對堿基排序),那么得到的測序結(jié)果是不到10億個(gè)短序列片段,一般每個(gè)短序列片段我們稱為讀取單位(reads)。
每個(gè)讀取單位含有100個(gè)堿基對(不同讀取單位的信息需要重疊,才能最后拼全),而每個(gè)堿基的錯(cuò)誤率范圍是0.1%到10%。所以,一直以來,對于HTS來說,最大的挑戰(zhàn)是把碎片化的結(jié)果信息拼成一整段完整的序列信息。
瓶中基因組聯(lián)盟Genome in a Bottle Consortium(GIAB),和精準(zhǔn)FDA平臺(美國藥監(jiān)局做的基因組信息學(xué)社區(qū)和共享數(shù)據(jù)平臺)一樣,致力于提高基于HTS基因測序結(jié)果。他們能提供高精度的人體基準(zhǔn)基因組序列信息。
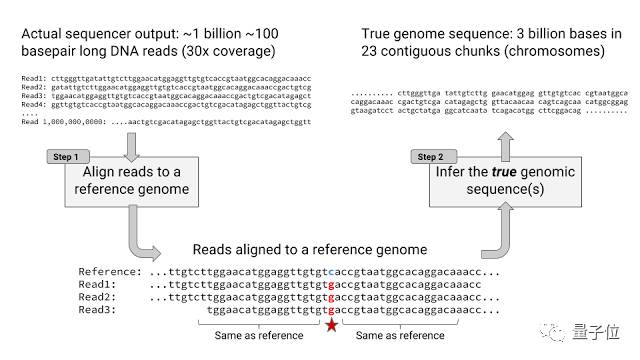
把測序結(jié)果與基準(zhǔn)基因序列一比對,就可以得到很多個(gè)堿基變異位點(diǎn)(就是上圖打星的地方),這些位點(diǎn),可能是SNP單核苷酸多態(tài)性導(dǎo)致的,也可能是測序過程中復(fù)制出錯(cuò)造成的。
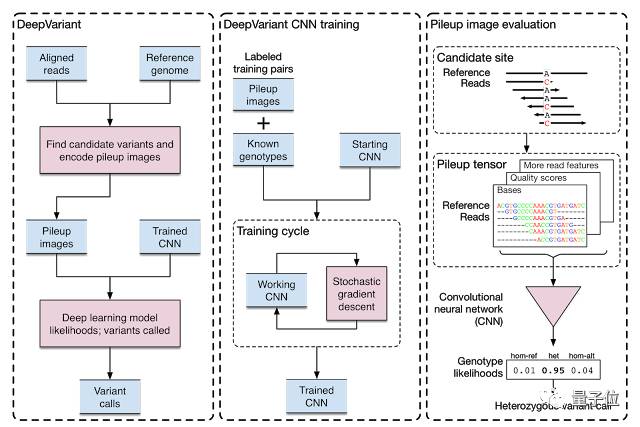
今天,Google Brain團(tuán)隊(duì),聯(lián)合同屬于Alphabet旗下的生命科學(xué)兄弟公司Verily,用了兩年多時(shí)間,研發(fā)出了一個(gè)名叫DeepVariant的開源工具,專門用深度神經(jīng)網(wǎng)絡(luò)來識別HTS結(jié)果中DNA測序數(shù)據(jù)里這些堿基變異位點(diǎn)。這個(gè)工具在準(zhǔn)確率上和精確度上,比傳統(tǒng)的比對拼接方法都高出一大截。
DeepVariant,把工作量巨大的拼接問題(HTS碎片化的結(jié)果拼接成完整的基因序列),轉(zhuǎn)變成了一個(gè)典型的圖像分類問題。而圖像分類正是谷歌擅長的技術(shù)。
2016年,DeepVarient還在PrecisionFDA Truth Challenge中贏得了最高SNP性能獎(jiǎng)(Highest SNP Performance)。在那之后,Google Brain團(tuán)隊(duì)又將錯(cuò)誤率降低了50%。
下面的四幅圖,分別代表實(shí)際測序的片段和基準(zhǔn)序列的比對結(jié)果。
△A:單核苷酸多態(tài)性造成的堿基變異位點(diǎn);
B:一條染色體上少了一個(gè)堿基;
C:兩條染色體上都少了一個(gè)堿基;
D:復(fù)制錯(cuò)了的堿基變異位點(diǎn)。
在比對過程中,要回答的一個(gè)關(guān)鍵的問題是,怎么判斷比對后得到的堿基變異位點(diǎn),是存在于兩條染色體中,還是只在一條里,還是都沒有。造成堿基變異位點(diǎn)的原因不只一種,最常見的三種可能是單核苷酸多態(tài)性,或多插了一個(gè)堿基,或少復(fù)制了一個(gè)堿基。
這些變異位點(diǎn)如果用視覺識別的算法就能快速找出來。大大提高HTS后的比對拼接的效率。
因?yàn)槠恐谢蚪M聯(lián)盟Genome in a Bottle Consortium(GIAB)提供的人體基準(zhǔn)基因組序列信息是高精度可信的,或者更嚴(yán)謹(jǐn)?shù)卣f,是最接近真實(shí)序列的信息。
通過這個(gè)基準(zhǔn)序列得到的復(fù)制數(shù)據(jù),谷歌團(tuán)隊(duì)可以拿它們來訓(xùn)練基于Tensor Flow的圖像分類模型,所得到的DeepVariant,最后可以區(qū)別真實(shí)序列數(shù)據(jù)和復(fù)制數(shù)據(jù)。
盡管DeepVariant根本不懂什么是基因組序列,也不懂HTS,但是只用了一年,就已經(jīng)贏得了PrecisionFDA Truth Challenge中的最高SNP性能獎(jiǎng)(Highest SNP Performance)。而且到目前為止,把已有最優(yōu)異的方法拼接錯(cuò)誤率降低了50%多。
在發(fā)布開源代碼的同時(shí),Google Brain還發(fā)布Google Cloud上的DeepVarient工作流,方便開發(fā)者用它來處理大型數(shù)據(jù)集。
最后,附上相關(guān)鏈接:
Google Research Blog介紹:https://research.googleblog.com/2017/12/deepvariant-highly-accurate-genomes.html
開源代碼:https://github.com/google/deepvariant
Google Cloud版:https://cloud.google.com/genomics/deepvariant
-
谷歌
+關(guān)注
關(guān)注
27文章
6173瀏覽量
105640 -
人工智能
+關(guān)注
關(guān)注
1792文章
47443瀏覽量
239024
原文標(biāo)題:谷歌推出開源工具DeepVariant,用深度學(xué)習(xí)識別基因變異
文章出處:【微信號:gh_ecbcc3b6eabf,微信公眾號:人工智能和機(jī)器人研究院】歡迎添加關(guān)注!文章轉(zhuǎn)載請注明出處。
發(fā)布評論請先 登錄
相關(guān)推薦




 谷歌推出開源工具DeepVariant,用深度神經(jīng)網(wǎng)絡(luò)識別基因變異
谷歌推出開源工具DeepVariant,用深度神經(jīng)網(wǎng)絡(luò)識別基因變異










評論