長久以來,在復(fù)雜、異質(zhì)的樣品中同時(shí)識(shí)別以及定位多個(gè)分子或者分子組裝的能力一直是推動(dòng)熒光顯微鏡在生物和物理科學(xué)研究中應(yīng)用的主要特性。例如,自1986年以來,使用四種光譜上的不同熒光團(tuán)來識(shí)別DNA中的腺嘌呤(A)、胸腺嘧啶(T)、胞嘧啶(C)和鳥嘌呤(G)堿基,這一直是大多數(shù)自動(dòng)化DNA測序技術(shù)的基礎(chǔ)。然而,對(duì)大規(guī)模生物系統(tǒng)的基因組和轉(zhuǎn)錄組的研究可能需要同時(shí)識(shí)別和定位成百上千的分子標(biāo)靶。這種高度并行的分析超出了基于光譜鑒別的多路復(fù)用能力。Lumencor分析了基于光譜鑒別的多路復(fù)用熒光檢測的局限性,以及為擴(kuò)大可檢測標(biāo)靶數(shù)量而引入的一些固態(tài)光源新技術(shù)。
光譜鑒別的局限性
大多數(shù)多路復(fù)用檢測方案都基于光譜鑒別,因?yàn)榕c基于時(shí)間或空間鑒別方法相比,它的技術(shù)復(fù)雜性較低,成本也較低。然而由于光譜串?dāng)_,光譜的鑒別方法范圍僅限于大約5個(gè)目標(biāo)(圖1和圖2)。這種局限性主要是由于用于雙分子標(biāo)記的熒光染料和熒光蛋白(FP)的光譜特性,如圖1 所示。在此示例中,雖然只有兩個(gè)熒光標(biāo)記,但它們的激發(fā)和發(fā)射波長跨越了整個(gè)可見光波長范圍(400-700nm),并且具有明顯的光譜重疊,會(huì)導(dǎo)致光譜分離不完全。如果以485nm左右的光進(jìn)行激發(fā)(灰色部分),兩種熒光團(tuán)會(huì)被同時(shí)激發(fā)。只有波長大于550nm時(shí)才能選擇性地激發(fā)其中的一種,從而獲得光譜鑒別。
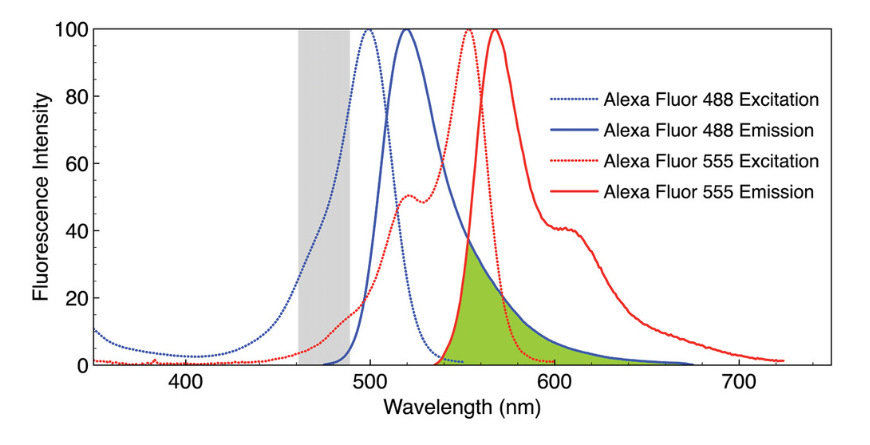
圖1. Alexa Flour488和Alexa Fluor 555熒光染料的歸一化熒光激發(fā)和發(fā)射光譜。發(fā)射光譜的重疊區(qū)域由綠色陰影表示。灰色陰影區(qū)域表示圖2中用于采集圖像A-C的激發(fā)帶寬(475/28nm)。
針對(duì)串?dāng)_的問題,雖然已經(jīng)開發(fā)出具有窄發(fā)射光譜的量子點(diǎn)納米晶體,可以提供更好的分離光譜。但與有機(jī)染料相比,這種改進(jìn)的代價(jià)是熒光團(tuán)尺寸增加了一個(gè)數(shù)量級(jí)以上,這反過來又阻礙了它們?cè)陔p分子標(biāo)記應(yīng)用中的應(yīng)用。
Lumencor的固態(tài)光引擎優(yōu)化了輸出光譜,提供了多個(gè)窄線寬的光源,盡可能實(shí)現(xiàn)對(duì)特定熒光染料的精確激發(fā),而這對(duì)于多重?zé)晒鈾z測至關(guān)重要,可以有效減小光譜串?dāng)_問題的發(fā)生。下圖中是使用Lumencor SPECTRA光引擎、Andor Zyla 5.5 sCMOS相機(jī)和Nikon Ti2顯微鏡對(duì)麂皮成纖維細(xì)胞進(jìn)行成像。可以看到由于激發(fā)光帶寬(555/28 nm)與Alexa Fluor 488的激發(fā)光譜沒有顯著交叉,因此圖像沒有因串?dāng)_而退化。
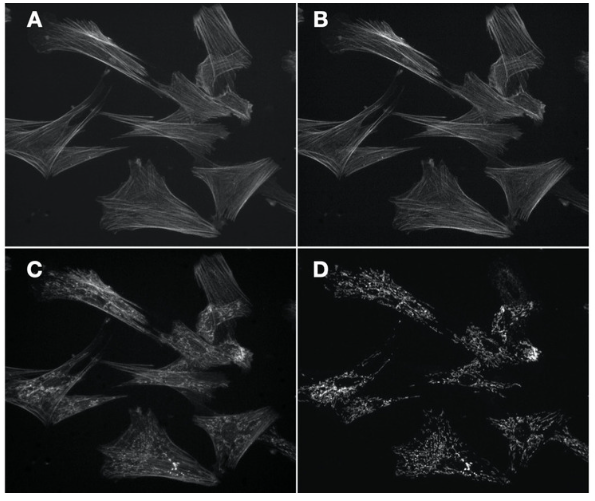
圖2. 四張60倍熒光圖像,展示了同一視野下的麂皮成纖維細(xì)胞。
用Alexa Fluor 488 phalloidin (actin)以及mouse anti–OxPhos Complex V + Alexa Fluor 555 goat anti–mouse IgG (mitochondria)進(jìn)行標(biāo)記。A.使用單波段激光和發(fā)散濾光片以及單波段的二色分光鏡來獲得的肌動(dòng)蛋白細(xì)胞骨架的圖像。B.使用單波段激發(fā)和發(fā)射濾光片以及多波段段二色分光鏡來獲得的等效肌動(dòng)蛋白細(xì)胞骨架圖像。C.由于光譜串?dāng)_導(dǎo)致線粒體檢測時(shí)肌動(dòng)蛋白細(xì)胞骨架圖像退化。圖像使用單波段激發(fā)濾光片、多波段二色分光鏡和多波段的發(fā)射濾光片獲得。D.使用單波段激發(fā)濾光片、多波段二色分光鏡和多波段發(fā)射濾光片獲得的線粒體圖像。
多重?zé)晒獬上竦耐黄?/strong>
好馬配好鞍,為了充分利用 Lumencor 光源的優(yōu)秀性能,科研人員正在進(jìn)一步開發(fā)和優(yōu)化用于多路復(fù)用熒光檢測的方案。
來自美國國家神經(jīng)系統(tǒng)疾病和卒中研究所的一篇Nature論文中有為解決多重?zé)晒獬上竦臒晒獯當(dāng)_問題提供了一些新策略。為了理解大腦中健康和病理生理過沖需要對(duì)多個(gè)組織樣本進(jìn)行大量的組織學(xué)觀察,以表征所有主要的腦細(xì)胞類型。為了實(shí)現(xiàn)大規(guī)模多重免疫熒光成像技術(shù),以實(shí)現(xiàn)多達(dá)10種不同熒光標(biāo)記物的同時(shí)成像,從硬件上,他們優(yōu)化了濾光片的組合,使用了10組自定義的激發(fā)/二向色/發(fā)射濾光片組合,這些精心挑選的濾光片被優(yōu)化用于迭代成像不同的光譜兼容的熒光標(biāo)記物,以確保在成像光譜范圍內(nèi)具有最小的光譜串?dāng)_。而從軟件上,本文中也開發(fā)了多種算法以從復(fù)雜的腦組織樣本中提取出豐富和準(zhǔn)確的生物學(xué)信息,主要是一種半監(jiān)督稀疏線性光譜解混算法,校正原始熒光圖像數(shù)據(jù)的串?dāng)_和非特異性標(biāo)記,提高了信號(hào)分離的準(zhǔn)確性。

Lumencor固態(tài)光引擎SPECTRA的光譜輸出針對(duì)本方案中使用的熒光染料面板的選擇性激發(fā)進(jìn)行了優(yōu)化,如圖3所示。光引擎集成了 LED、光管和激光器,以實(shí)現(xiàn)所需的光譜激發(fā)分布和所需的光功率。使用相同的熒光染料組合和濾光片配置,通過連續(xù)的剝離、重新標(biāo)記和成像輪次,可以增加多重檢測的程度。通過5個(gè)周期的剝離、10倍多重標(biāo)記和成像,已證明可以在大鼠大腦冠狀切片中同時(shí)定位50個(gè)生物標(biāo)記靶標(biāo)。
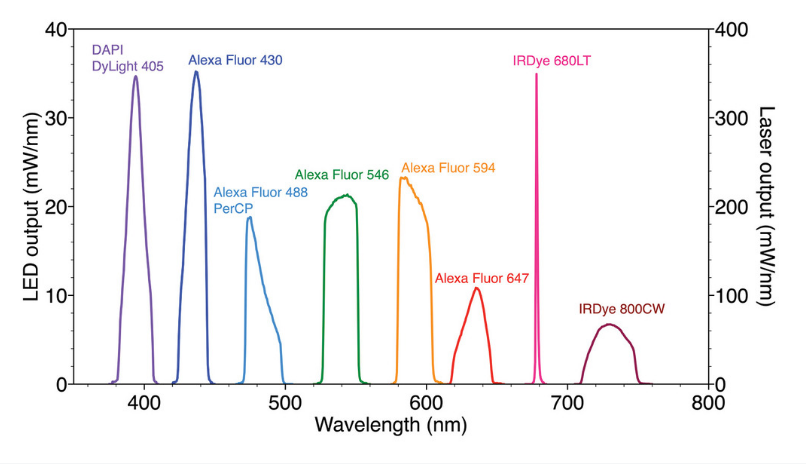
圖3. SPECTRA光引擎的光譜輸出經(jīng)過優(yōu)化,可選擇性激發(fā)用于10倍免疫熒光檢測的熒光染料。
參考文獻(xiàn):
D Maric, J Jahanipour, B Roysam et al., Whole-brain tissue mapping toolkit using large-scale highly multiplexed immunofluorescence imaging and deep neural networks. Nature Communications (2021)12:1550
MERFISH技術(shù)
通過在基于光譜鑒別的多路復(fù)用技術(shù)中添加空間和時(shí)間維度,已經(jīng)開發(fā)出能夠同時(shí)識(shí)別和定位數(shù)百和數(shù)千個(gè)生物分子標(biāo)靶的多路復(fù)用技術(shù)。而用于基因表達(dá)分析的分子條形碼掃描就是這樣一種技術(shù),利用核酸雜交來識(shí)別DNA和RNA分子標(biāo)靶,并將其作為產(chǎn)生熒光染料順序排列的模版。
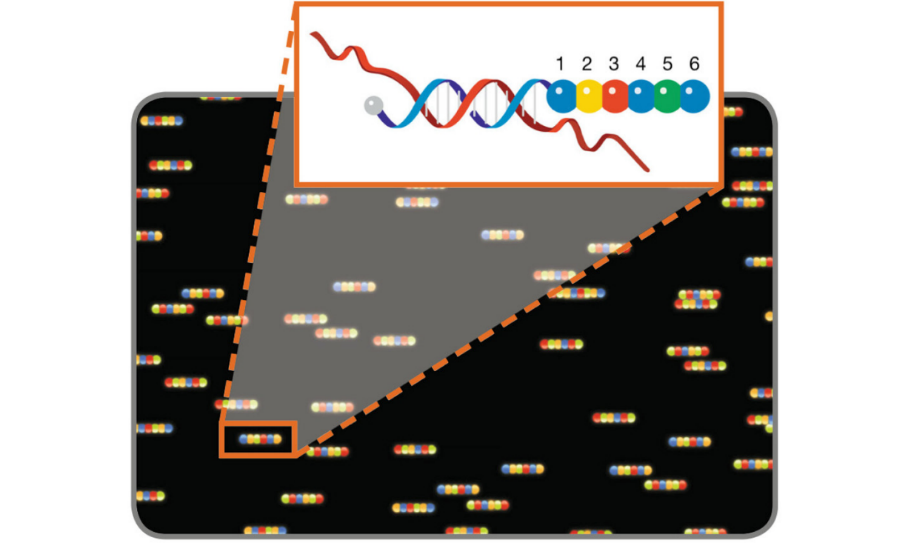
圖4. 用于基因表達(dá)分析的分子條形碼掃描。掃描儀配有激發(fā)和發(fā)射濾光片,可區(qū)分由藍(lán)色、綠色、黃色和紅色圓圈代表的 4 種染料,可讀取探針位置 1 至 6 的熒光信號(hào),以揭示雜交核酸靶標(biāo)的編碼身份。
以分子條形碼掃描為出發(fā)點(diǎn)的MERFISH (多重誤差魯棒熒光原位雜交)是一種高度多重化的單分子成像技術(shù),能夠同時(shí)測量單個(gè)細(xì)胞中數(shù)百至數(shù)千個(gè)目標(biāo) RNA 種類的拷貝數(shù)和空間分布。首先將編碼的寡核苷酸探針和目標(biāo)RNA雜交,并依次添加熒光染料標(biāo)記的讀取核苷酸探針。這些讀取探針會(huì)在單分子級(jí)別上被空間定位,然后被熄滅。讀取探針必須僅與編碼探針雜交,而不能與細(xì)胞核酸發(fā)生雜交。通過連續(xù)的成像周期積累編碼探針的身份,如果編碼探針通過讀取探針的雜交被檢測到,則賦予二進(jìn)制位值“1”,如果沒有檢測到,則賦予“0”。原則上,需要 12 個(gè)雜交+成像周期來識(shí)別 4095(212-1)個(gè)獨(dú)特的編碼探針。實(shí)際上,二進(jìn)制編碼空間較少,以盡量減少假陰性(1>0)和假陽性(0>1)調(diào)用錯(cuò)誤以及隨之而來的靶標(biāo) RNA 錯(cuò)誤識(shí)別。例如,已經(jīng)證明可以使用 69 位條形碼識(shí)別 10,050 個(gè) RNA 靶標(biāo)。
對(duì)于MERFISH應(yīng)用,需要高強(qiáng)度、空間均勻的照明場,并且與典型sCMOS相機(jī)傳感器的尺寸(~200mm2)相匹配。為了滿足此要求,需要在顯微鏡中安裝專業(yè)的臨界照明器。Lumencor的CELESTA光引擎很好地滿足了這些需求,通過光纖輸出耦合到顯微鏡中,通過高數(shù)值孔徑的高放大倍數(shù)物鏡在樣品平面上提供1-10W/mm2。
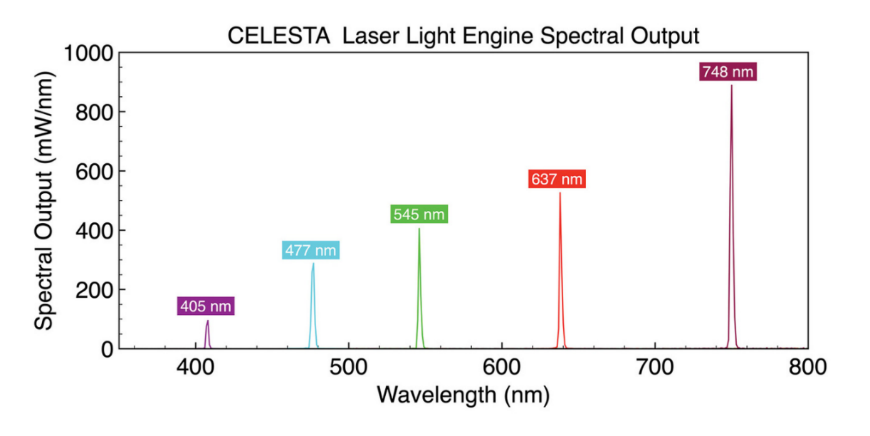
圖5. 為MERFISH多路單分子成像優(yōu)化的CELESTA激光光引擎輸出光譜。
顯然,對(duì)于那些超出簡單光譜分辨能力的高并行熒光標(biāo)記生物分析,可以通過精心選擇熒光團(tuán)、光學(xué)濾光片、精細(xì)調(diào)節(jié)激發(fā)照明和智能算法來實(shí)現(xiàn)。巧妙的標(biāo)記策略,如分子條形碼,也可以應(yīng)用于廣泛的生物樣本,以增強(qiáng)來自高度復(fù)雜組織標(biāo)本的生物分子信息的光譜分辨和空間分布。在此,我們講述了一種大規(guī)模并行單分子成像技術(shù),MERFISH,利用重復(fù)標(biāo)記協(xié)議來最大化單細(xì)胞中數(shù)十萬個(gè)RNA靶標(biāo)的拷貝數(shù)量和空間分布信息。檢索大量數(shù)據(jù)集的技術(shù)和機(jī)會(huì)對(duì)于當(dāng)今豐富的基因組和轉(zhuǎn)錄組信息生物分析至關(guān)重要。實(shí)驗(yàn)者必須在有限的波長范圍內(nèi)進(jìn)行光譜解析,以充分利用光譜寬度、光譜純度和熒光團(tuán)兼容性,以實(shí)現(xiàn)激發(fā)和發(fā)射之間的最佳區(qū)分。策略性的重復(fù)成像,輔以純凈、明亮、穩(wěn)定的照明和高質(zhì)量的光學(xué)玻璃,能夠?qū)崿F(xiàn)高度多重化的結(jié)果,并克服了在可用可見波長內(nèi)定義的歷史限制。
-
固態(tài)照明
+關(guān)注
關(guān)注
1文章
77瀏覽量
15601 -
顯微鏡
+關(guān)注
關(guān)注
0文章
629瀏覽量
24330 -
熒光檢測
+關(guān)注
關(guān)注
0文章
7瀏覽量
6633
發(fā)布評(píng)論請(qǐng)先 登錄
NMUX1237模擬多路復(fù)用器/解復(fù)用器規(guī)格書

CBT3251 FET多路復(fù)用器/多路分解器規(guī)格書

CBT3253 FET多路復(fù)用器/多路分解器規(guī)格書

CBT3257A多路復(fù)用器/解復(fù)用器規(guī)格書

在多路復(fù)用模擬輸入應(yīng)用中使用ADS8411

AM18xx引腳多路復(fù)用實(shí)用程序

AM17xx引腳多路復(fù)用實(shí)用程序

通過使用多路復(fù)用器實(shí)現(xiàn)基于SPI的閃存擴(kuò)展

多路復(fù)用器應(yīng)用中的防護(hù)

基于帶寬的無源多路復(fù)用器>Ron

電源多路復(fù)用器基礎(chǔ)知識(shí)

為MCU擴(kuò)展選擇正確的多路復(fù)用器

如何使用多路復(fù)用器處理高壓共模應(yīng)用

多路復(fù)用器將取代繼電器應(yīng)用說明

多路復(fù)用器和信號(hào)開關(guān)詞匯表




















評(píng)論