近日,天津大學精密儀器與光電子工程學院的光子芯片實驗室提出了一種基于深度學習的二維拉曼光譜算法,成果以“Rapid and accurate bacteria identification through deep-learning-based two-dimensional Raman spectroscopy”為題,發表在《Analytica Chimica Acta》上。
表面增強拉曼光譜(Surface-enhanced Raman spectroscopy, SERS)可以提供分子獨特的振動指紋,具有預處理簡單、靈敏度高和無創檢測等優點。隨著人工智能(artificial intelligence, AI)技術的快速發展,基于AI的拉曼光譜分析在細菌識別等生物傳感領域具有廣闊前景。目前,為了更加精確地實現細菌識別,研究者們提出將一維拉曼光譜轉換為二維拉曼光譜圖來獲取更加豐富的數據信息。但是,高分辨率的二維拉曼光譜圖在提高識別準確度的同時,通常會引起計算時間過長等問題。
在本項工作中,研究者們提出了一種低分辨率、多信息量的二維拉曼光譜圖處理算法,驗證了其在細菌識別中的可行性。該算法基于小波包變換與格拉姆角場(Wavelet Packet transform and Gramian Angular field, WPGA)技術,利用深度學習模型實現快速高效地識別細菌。如圖1所示,識別過程主要包括三個步驟,首先,采集細菌樣品的SERS光譜并進行預處理;其次,利用WPGA算法將一維拉曼光譜轉換成二維拉曼光譜圖,這是本項工作能夠實現快速高效分類的關鍵。由于分子振動,拉曼光譜常會出現微小的特征峰,但由于這些信息頻率相對較高,在光譜平滑過程中容易被忽略。因此,我們采用小波包變換技術,對高頻信息成分進行更為精細的篩選,以減少有用信息的損失。然后利用格拉姆角場技術,對篩選后的有效特征添加序列信息來提高識別精確度。最后,通過訓練深度學習模型來識別細菌。
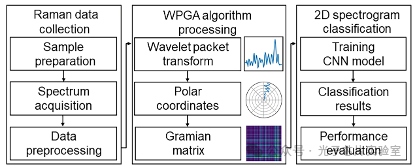
圖1.細菌識別的主要步驟。包括三個步驟:拉曼數據收集、WPGA算法處理和二維光譜圖分類。
基于提出的算法,研究者們對枯草芽孢桿菌和酵母菌的SERS光譜進行了分類驗證。圖2(a)展示了基于卷積神經網絡進行該二元分類問題的訓練過程,結果表明驗證集的精度可以達到99.75%,損失為0.0074。圖2(b)展示了模型訓練完成后,使用測試集來評估分類性能,識別準確率為99.64%,初步證明了該算法的可行性。為了驗證該算法的泛化能力,研究者們進一步開展了基于Bacteria-ID公共數據集的多分類識別任務。圖2(c)展示了沒有進行WPGA算法處理時,30種細菌在二維空間中的分布,不同類別之間的相互混合,難以區分。如圖2(d)所示,利用所提算法后,不同種類的細菌彼此分離,基于Resnet網絡模型的分類識別準確率可以達到90.55%。
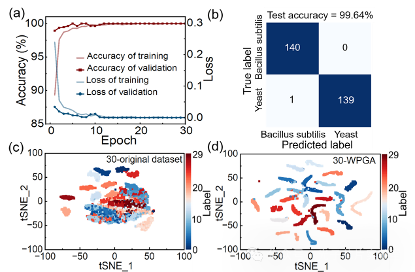
圖2.兩種及多種細菌的分類識別結果。(a)訓練集和驗證集的準確率和損失曲線;(b)測試集的混淆矩陣;(c)數據集在二維空間中的分布;(d)使用WPGA算法后,數據集在二維空間中的聚類效果。
本論文第一作者為天津大學精密儀器與光電子工程學院的碩士生劉怡辰,通信作者為天津大學精密儀器與光電子工程學院的程振洲教授、胡浩豐教授和高翊盛博士。該工作得到了國家自然科學基金(62161160335、62175179、62475188、61805175、12304428)、天津市自然科學基金(23JCJQJC00250)、廣東省自然科學基金(2022B1515130002)和日本學術振興會(JP18K13798)項目的支持。
-
算法
+關注
關注
23文章
4607瀏覽量
92829 -
二維
+關注
關注
0文章
40瀏覽量
11983 -
深度學習
+關注
關注
73文章
5500瀏覽量
121111 -
拉曼光譜
+關注
關注
0文章
83瀏覽量
2738
原文標題:面向細菌識別應用的二維拉曼光譜處理算法
文章出處:【微信號:光子芯片實驗室,微信公眾號:光子芯片實驗室】歡迎添加關注!文章轉載請注明出處。
發布評論請先 登錄
相關推薦
一種基于二維離散小波變換的醫學圖像增強算法
廣義二維相關光譜學發展
一種快速二維到來方向估計算法
基于深度學習的二維人體姿態估計算法

從單層石墨烯中收集拉曼光譜





 一種基于深度學習的二維拉曼光譜算法
一種基于深度學習的二維拉曼光譜算法















評論