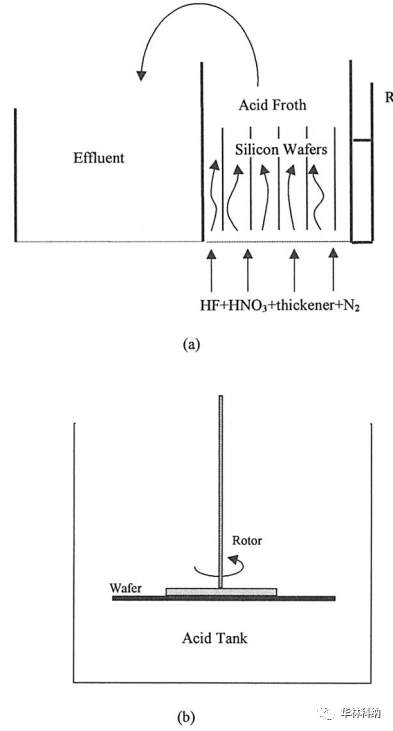
自誕生以來,GPU就被廣泛應用于高性能科學計算領域。GPU的大規模并行計算能力幫助包括分子模擬在內的許多傳統科學計算應用實現了爆炸式提速。分子模擬是一種利用計算機在微觀尺度下研究材料、蛋白質、藥物等分子行為的技術。由于其模擬流程中包含許多高計算量的模塊,分子模擬程序非常適合通過GPU來加速。經過十幾年的發展迭代,目前大部分常用的分子模擬軟件都對GPU硬件進行了不同程度的適配。本文將對分子模擬中關鍵的近程力計算模塊在三款不同模擬軟件中的GPU移植案例展開深度剖析,希望能以小窺大,探究科學計算應用在GPU上優化的關鍵點。
在本系列的上篇《分子模擬:打開微觀世界的鑰匙(上)—— 起源、發展與現狀》,我們介紹了分子模擬技術的起源、發展與現狀。分子模擬技術可以通過一個體系的化學組成和其所處的環境來預測其物理/化學以及動力學性質,是科學計算領域的一個重要組成部分。而作為一項歷史悠久的仿真技術,傳統的分子模擬程序通常都是通過多線程的方法在CPU上進行模擬。但在GPU橫空出世以后,人們驚喜地發現,在GPU的超高并行效率的協助下,分子模擬的速度和模擬體系的尺寸都可以得到數量級的提升。因此,目前的大部分主流分子模擬應用中都加入了GPU版本供用戶使用。然而,仔細研究后可以發現,各大應用在GPU上的適配方法差異很大。本文將以分子動力學計算中的近程力計算模塊(short-range force)為例,拆解幾大主流應用中的GPU算法細節。本文中提到的GPU術語皆以CUDA體系為標準,包括:thread(線程), warp(線程束), shared memory(共享內存), host(主設備), device(計算設備)等。
1
分子動力學近程作用力計算模塊
本系列上篇曾經介紹過,分子動力學模擬中體系力場的計算可以被大致分為兩大部分:成鍵作用力(bonded interactions)和非成鍵作用力(nonbonded interactions)。顧名思義,成鍵作用力計算的是互相形成化學鍵的原子之間的作用力,而非成鍵作用力則是體系中所有沒有形成化學鍵的原子間的相互作用力。由于非成鍵作用力的計算需要遍歷體系中所有可能的原子對,其復雜度達到了 O(N2) (此處N為體系中的總原子數)。因此,非成鍵作用力的計算是分子動力學模擬中最耗時的部分。考慮到該作用力隨距離的增加而快速衰減的特性,我們可以通過引入截斷距離(cutoff distance)的概念來簡化計算。我們將小于截斷距離的部分作用力稱為近程作用力,而大于截斷距離的部分則稱遠程作用力。
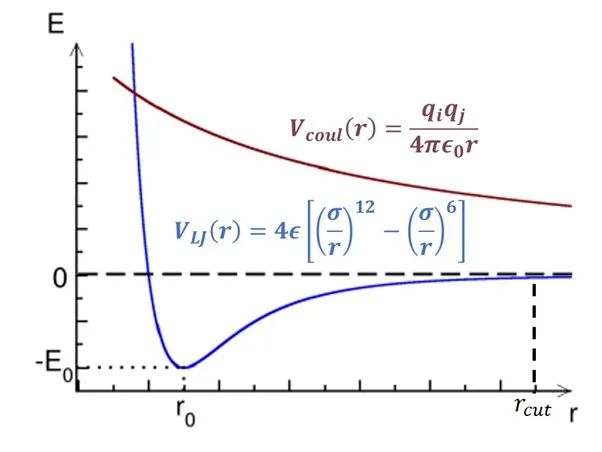
圖1:非成鍵作用勢隨距離變化簡圖。
橫軸為距離,縱軸為勢能。
如圖1所示,范德華勢(VLJ, 色散作用)和庫倫勢(Vcoul, 電荷作用)都隨距離r的增加而遞減。其中范德華勢在截斷距離(rcut)處基本衰減到零,因此大部分情況下這部分計算只需要考慮截斷距離內的原子對。但庫倫勢衰減速度較慢,我們無法直接忽略其長程(>rcut)部分。庫倫勢的長程部分可以通過目前最先進的PME(particle-mesh Ewald)方法[1]計算,該算法的時間復雜度僅為O(NlogN)。由此可見,近程作用力的計算(O(N2))是模擬體系力場算法中最消耗計算資源的部分。如圖2所示,非成鍵作用力中的近程力計算開銷(SR+NEIGH)在總開銷中的占比高達~80%。因此,近程力計算模塊的加速是提升模擬速度的關鍵所在,該模塊也成為了最早被移植到GPU上的計算單元,目前各大主流模擬軟件中都有較為成熟的GPU近程力計算版本。后面幾個小節中,筆者將討論近程力計算模塊在CPU上和不同軟件的GPU版本中的算法差異。
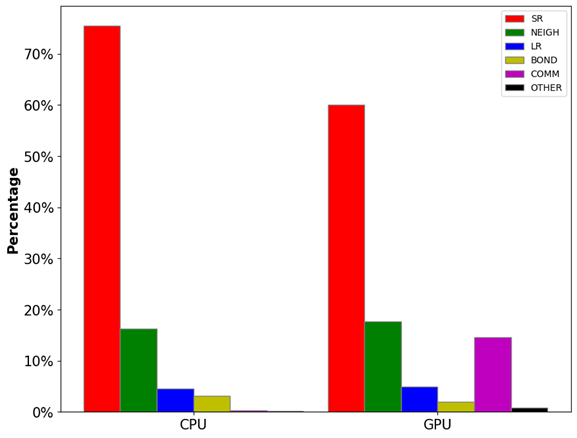
圖2:LAMMPS分子動力學模擬中各模塊的計算開銷占比。其中,SR為近程力計算開銷,NEIGH為鄰近列表構建開銷,LR為長程力計算開銷,BOND為成鍵作用力計算開銷,COMM為通訊開銷,OTHER為其他開銷。
測試體系:視紫紅質(Rhodopsin)蛋白質體系,96,000原子,LAMMPS Benchmarks
2
近程力CPU算法
對于一個包含N個原子的體系,計算近程作用力最直接的方法就是雙循環遍歷所有可能的原子對,并通過距離和力場參數計算部分作用力,最后對所有結果進行歸約操作。很明顯,即使在引入截斷距離以后,此種暴力求解方法的時間復雜度仍為O(N2)。因此,我們需要一些技巧來加速近程力計算模塊,而neighbor list(鄰近列表)方法就是一種最常用的技巧。neighbor list方法為體系內的每一個原子都構建一張列表,其中存儲著在三維空間中與其臨近(距離小于截斷距離)的其他原子的編號。當這些列表構建完畢以后,在計算近程力時,我們只需要遍歷每個原子與其neighbor list中的其他原子的作用力即可,因為截斷距離外的作用力可以忽略不計。如此一來,近程力計算的復雜度就被減小到了O(N)。
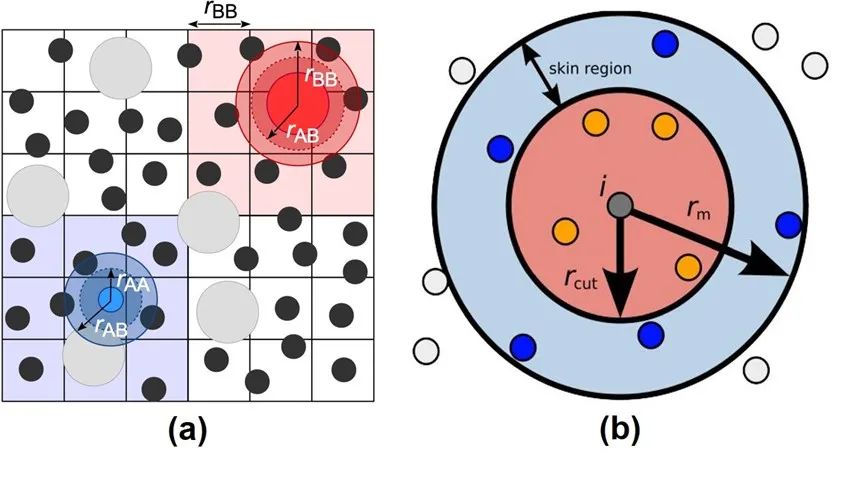
圖3:Verlet Neighbor List方法示意圖。
來源:Neighbor lists — HOOMD-blue 2.5.2 documentation
然而,構建neighbor list的時間是無法被忽略的。同時,由于在模擬過程中原子位置會持續發生變化,這些列表需要頻繁地被重建。于是,人們提出了一種可以將neighbor list重復使用的技巧: Verlet neighbor list方法[2]。考慮到原子運動的連續性以及分子動力學模擬中通常步長很短等特性,原子的坐標在若干步模擬過后通常不會改變太大。因此,如圖3(a)所示,我們可以把模擬體系劃分成若干網格, 將每一個原子歸到格子里, 然后只需要搜尋原子當前以及周圍總共27個(假設3D體系)網格中的其他原子即可。另外,為了記錄原子進入或離開neighbor list范圍的情況,我們還可以在截斷范圍以外設置一個緩沖區域(skin region), 如圖3(b)所示。假如監測到有大量原子離開緩沖區,則需立刻重構Verlet list;否則,可以按一定的頻率(一般每20到40步模擬)來更新list。如此便可以做到Verlet list的復用,節省大量計算開銷。圖4展示了CPU中近程力計算的兩大模塊,neighbor list build (a)和粒子間作用力計算(b)的簡單計算流程。


圖4:近程力CPU計算模塊:
(a) neighbor list 構建, (b) 作用力計算。
3
近程力LAMMPS-GPU算法
本文后面的篇幅中,我將介紹三款分子動力學應用中的近程力GPU算法。首先要介紹的是LAMMPS(Large-scale Atomic/Molecular Massively Parallel Simulator)[3]。LAMMPS 是一款由美國桑迪亞國家實驗室(Sandia National Lab)開發的開源分子動力學模擬程序包。該程序于1995年第一次發布,距今已有將近30年的歷史。
LAMMPS的GPU包中對近程力模塊的移植相對較為直接,并沒有對數據結構進行大幅改變,只是在最新的版本中加入了更多warp-level指令的使用。首先,LAMMPS-GPU的粒子間作用力的計算基本與CPU版本(見圖4(b))完全一致。簡單來說,即GPU中每一個thread負責一個原子,循環計算該原子與其neighbor list內的原子的相互作用力,再將結果寫入到儲存作用力的數組中。而在neighbor list構建模塊中,LAMMPS的GPU版本與CPU版本最大不同是通過warp shuffle的指令來減少從host到device的數據讀取。具體來說,該算法分為以下步驟:

圖5:LAMMPS-GPU neighbor list build模塊計算流程。
得益于GPU的高度并行能力以及warp 內部的數據共享能力,LAMMPS的GPU版本確實比CPU提升了不少速度。然而,這種直接的GPU移植方式還是存在著許多的缺陷,包括:
01
沒有改變傳統neighbor list的結構,LAMMPS的neighbor list 數據結構如圖6所示,每個粒子都有一個自己的neighbor list,導致有很多的neighbors被重復儲存。使用這樣的數據結構會大幅增加host-device間的數據傳輸壓力。
02
網格是通過空間坐標均勻劃分的,當處理非均質體系時各warp需要處理的粒子數差異較大,易造成線程分歧(thread divergence)。
03
計算粒子間作用力時沒有利用好shared memory/warp shuffle功能來減少從host到device的數據傳輸。
由此,我們可以看到,想要更好地激發GPU的潛力,程序還需要對底層的數據結構和運算邏輯進行進一步優化。
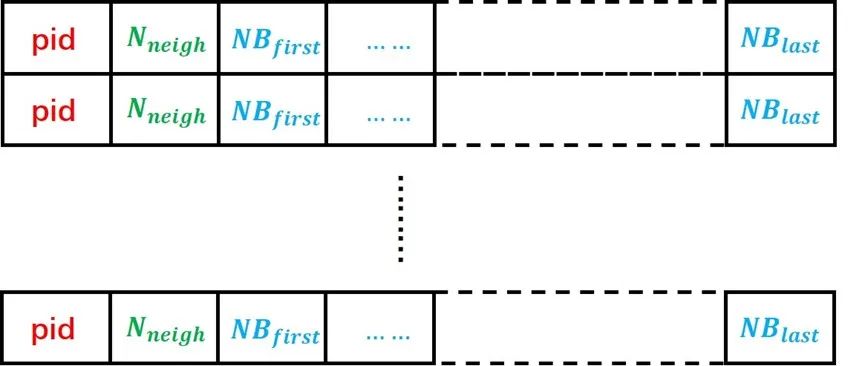
圖6:LAMMPS中的neighbor list數據結構。每個原子對應其中的一行數據,pid為原子編號,Nneigh為該原子的neighbor總數,NBfirst至NBlast為從第一個neighbor到最后一個neighbor的編號。
4
近程力GROMACS-GPU算法
GROMACS(GROningen MAchine for Chemical Simulations)[4]是一款由荷蘭格羅寧根大學團隊開發的模擬程序,該軟件于1991年首次發布,目前已經成為全球使用人數最多的分子動力學軟件之一。GROMACS從2013年的4.5版本開始支持GPU,經過多次迭代優化之后,目前的最新版本已經可以在GPU上完成全模擬流程。
與LAMMPS-GPU不同的是,GROMACS的開發團隊意識到了傳統的neighbor list結構不適合GPU的SIMT(單指令多線程)計算特性。因此,他們提出了一種基于cluster(聚類)結構的新型近程力算法[5]。受到矩陣乘法運算的啟發,該方法通過將體系中的粒子分為N x M大小的cluster來提升GPU的計算效率。
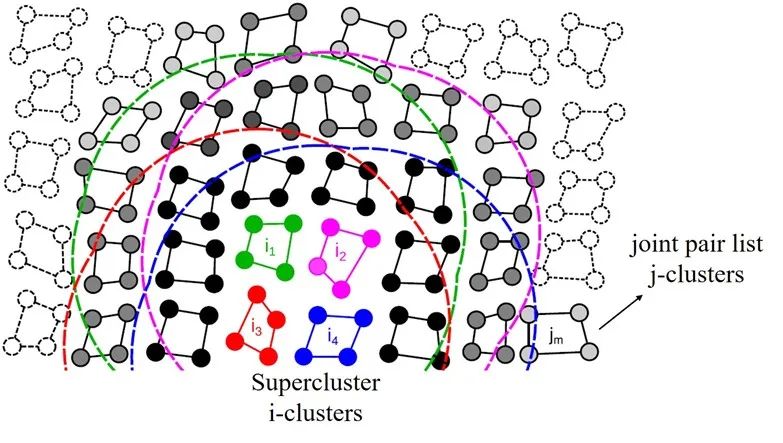
圖7:GROMACS-GPU算法中的super-cluster結構。圖中的super-cluster包含i1-i4 四個i-cluster,這些i-cluster的neighbors被整合成了一個joint pair list, 即圖中黑色和灰色的j-cluster合集。
來源:Páll etc. “Heterogeneous parallelization and accelerationof molecular dynamics simulations in GROMACS”, J. Chem. Phys. 153, 134110 (2020)
以圖7為例,圖中的體系被分為包含N=4個粒子的i-cluster和包含M=4個粒子的j-cluster。不同于以單個粒子為計算單元的傳統neighbor list算法,此新方法以粒子的聚類為單位來構建neighbor list。具體來說,在搜索neighbor的過程中,程序會遍歷所有的i-cluster,并找到在其截斷距離范圍內的j-cluster加入其neighbor list。如此一來,在計算作用力的時候就可以每次同時載入M?個粒子的信息,從而減少對neighbor數據的重復讀取操作。盡管這種做法會無可避免的增加一些無效計算(并不是cluster中的每個粒子都在目標粒子的截斷范圍內),但是對于這樣一個瓶頸在內存操作的程序來說,使用該方法的收益還是遠遠大于損失。
在實際應用中,這種聚類臨近列表(cluster neighbor list)方法可以進行進一步的優化來占滿目標硬件的執行寬度(execution width)。以Nvidia的GPU產品為例,N卡以warp為計算單元,每個warp中包含32個thread,因此其執行寬度為32。我們可以通過將cluster的大小設置為N=8,M=4 來達到最高計算效率。同時,還可以將4個i-cluster合并成一個super-cluster(見圖7中的i1-i4), 并將它們的neighbor list整合成一個聯合成對列表(joint pair list)。如此操作的好處在于可以充分利用GPU中較為快速的shared memory來進一步減少host到device端的數據傳輸。在每一次計算作用力時,程序會先將super-cluster中所有粒子的數據讀取進來并儲存在shared memory中,這樣就避免了在后續的計算過程中進行緩慢的顯存讀取(global memory load)操作。同理,在執行寬度為64的AMD GPU上,需要設置N=M=8 ; 而在英特爾硬件上則需將cluster的大小設置為N=4,M=2 。
通過對底層數據結構的改進優化,GROMACS成功提升了GPU計算資源的利用效率。與傳統的neighbor list結構相比,cluster pair list的使用可以顯著減少同一個neighbor的重復儲存,在數據復用和內存使用等方面做到了很好的優化。同時,相對均勻的計算分配也可以大幅縮短thread的閑置和等待時間。盡管增加了一定的計算量,但是在這個內存瓶頸的程序中,這部分增加的計算時間可以被隱藏在內存操作延遲中,從而不對整體性能產生很大影響。因此,筆者認為GROMACS-GPU的近程力計算模塊是一個很成功的GPU移植和優化案例。
5
近程力OpenMM-GPU算法
接下來要介紹的OpenMM[6]是一款主打易用性、拓展性和高性能的新型模擬引擎。該軟件的開發團隊主要來自斯坦福大學,他們設計這款軟件的初衷就是想要解決傳統分子模擬軟件難上手、源碼修改困難以及在GPU上性能不佳等問題。經過近十年的努力,OpenMM也確實越來越受學者們的歡迎。一方面,OpenMM的Python 和C++ API都做的非常完善,用戶可以自由添加新功能或者修改現有功能。另一方面,OpenMM的GPU算法先進,加速效果非常好。因此,許多新的研究項目都選擇其作為模擬引擎,包括著名的蛋白質結構預測模型Alpha Fold[7]。
為了提升近程力計算在GPU上的性能,OpenMM同樣在neighbor list的數據結構上進行了優化。其整體的優化思路與GROMACS比較類似,也是通過將多個粒子集合成atom block(原子塊)的方式來構建neighbor list,以減少neighbors的重復儲存和數據傳輸。兩種方法的最大區別在于每個集群中粒子數量的選擇。OpenMM的atom block中包含32或64個原子,而GROMACS中的cluster則只有4或8個粒子。增加集群的尺寸可以減少neighbor list搜索和構建的開銷,因為這部分計算的耗時是由體系中的集群總數決定的。但同時,大集群也意味著在作用力計算模塊中會引入更多的無效計算,增加計算資源的浪費。因此,集群尺寸的選取實際上是近程力計算中兩大模塊之間的平衡取舍問題。OpenMM團隊經過驗證后發現較大的集群在現代GPU架構中可以更為高效。同時,他們還使用了一些技巧來降低增大集群尺寸后引入的負面效應。比如,在構建neighbor list的時候增加一個細致搜索的步驟。
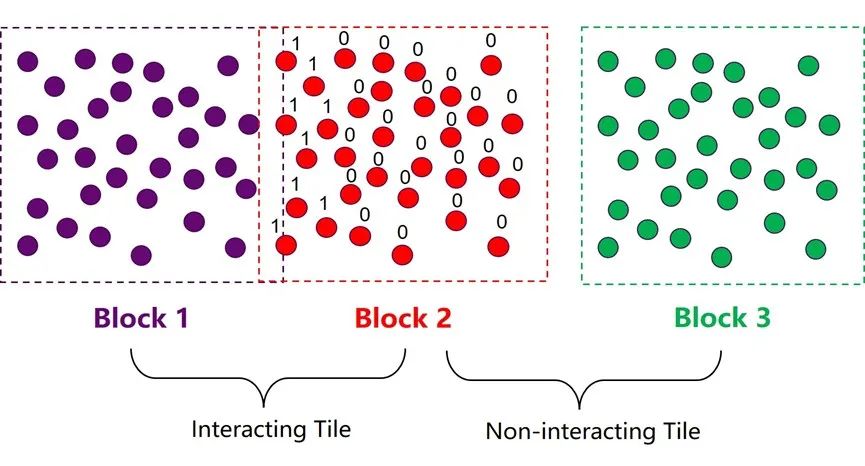
圖8:OpenMM-GPU算法中的atom block 和tile結構。
如圖8所示,在第一步粗略搜索的過程中,程序會將邊界有交集的兩個atom block 標記為相互作用集群對(interacting tile);邊界沒有交集的atom block對則被標記為互不作用集群對 (non-interacting tile)。這一步搜索結束以后,程序會馬上進入下一步細致搜索階段,這一步的目的是構建一個32/64位的mask,對block 2中的所有粒子進行判定,假如一個粒子與block 1中的任意粒子有相互作用,則將該位設為1,反之則設為0。這個mask中儲存的信息可以有效規避后續作用力模塊中的大部分冗余計算。OpenMM中這種類似的小trick還有很多,正是因為他們對GPU更深的理解和對細節的注重,使得OpenMM在GPU上的加速效果達到了業界最頂尖的水平。
結語
本文介紹了三款常用的分子動力學模擬應用中的GPU加速算法,分別代表三種不同程度的GPU適配思路。其中,LAMMPS的GPU版本基本直接沿用了CPU上的算法,只是利用GPU的高度并行能力對其中的計算密集部分進行加速。而GROMACS和OpenMM則將CPU版本中的底層數據結構和計算邏輯進行了大刀闊斧地改造,以更好地發揮GPU的硬件特性。另外,OpenMM通過一些算法細節上的巧妙設計進一步提升了程序在GPU上的運行效率。
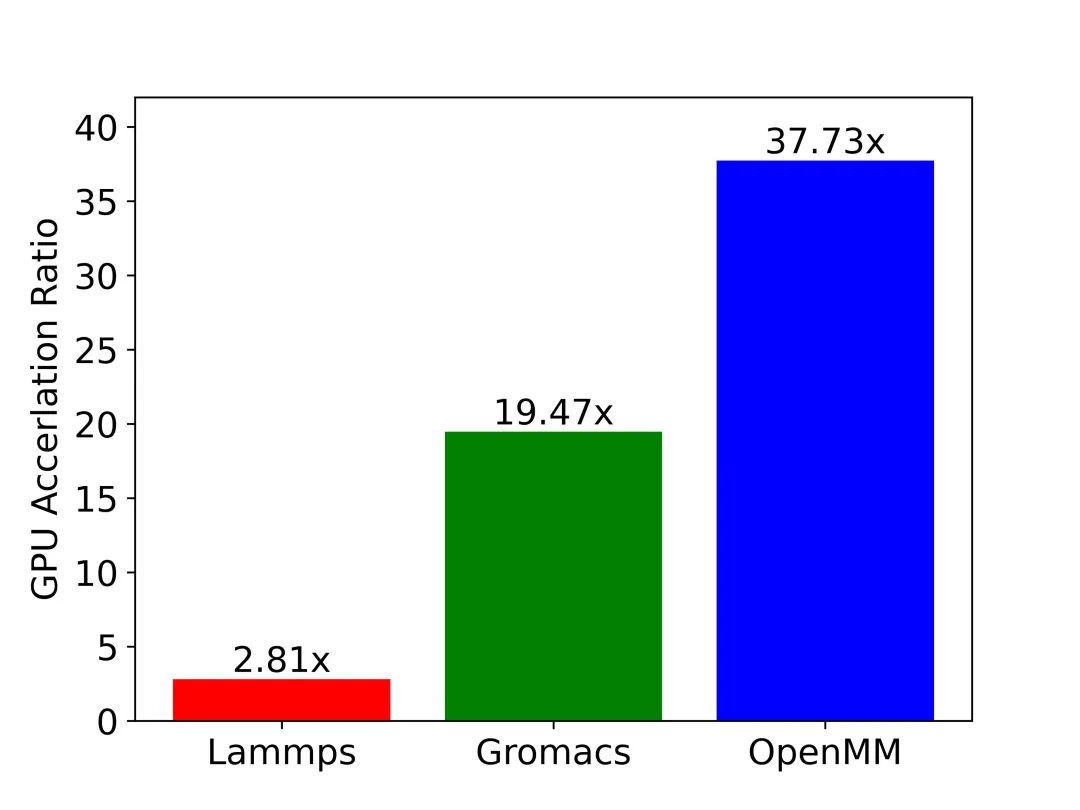
圖9:三大應用GPU加速效果一覽。圖中縱軸為GPU相對CPU的加速比。測試用例:包含45,000原子的SPC/E模型水體系,通過沐曦MetaMol分子模擬工作流軟件實現相同體系在不同應用上的模擬。
測試硬件:CPU: AMD Ryzen 7 3700X 8-Core Processor; ?GPU: NVIDIA GeForce RTX 3080。
圖9中展示了LAMMPS、GROMACS和OpenMM在模擬SPC/E模型水體系時的GPU-CPU的加速比數據。測試結果與前文中的分析一致:在相同的硬件上模擬同一體系時,OpenMM的GPU加速效率要遠高于GROMACS和LAMMPS。讀到這里大家應該都有疑問:為什么LAMMPS不對數據結構進行優化呢?究其原因,以LAMMPS為代表的傳統軟件歷史悠久,在設計之初并沒有考慮到在不同平臺、不同硬件上適配的需求,導致軟件的移植工作量巨大,而修改底層數據結構更是牽一發而動全身,幾乎難以完成。另一方面,像OpenMM這種近年來開發的新型應用則對軟件的拓展性和靈活性非常重視。比如在OpenMM中,用戶可以很便捷地通過插件的形式來添加新的計算平臺或是新算法。因此,在各種硬件架構層出不窮,日新月異的今天,如何保證拓展性和靈活性需要成為我們開發科學計算軟件時必須考慮的問題。
個人認為這些軟件的GPU移植和優化的實例比較具有研究價值,希望這篇文章可以給各位讀者帶來一些啟發,拓寬一些思路。本文中的大部分內容都來自筆者自己對軟件源碼的研究和模擬數據的分析所得。文中提出的觀點都來自我的個人理解,難免有所紕漏,歡迎大家批評指正。最后,希望通過進一步的優化和改進,我們可以讓分子模擬在沐曦的通用GPU上達到更高的速度和精度,真正成為人類打開微觀世界的鑰匙。
編輯:黃飛
 電子發燒友App
電子發燒友App
























評論